Actualités
3 actualités
Découvrez la série d’articles sur le thème « Les Femmes au Cœur de la Science à l’US2B ».
Les algues brunes sont une espèce clé dans la protection des écosystèmes côtiers aujourd’hui gravement menacées par le changement climatique. Une équipe de scientifiques, à laquelle ont été associés des chercheuses et chercheurs de l’Unité des Sciences en Biologie et Biotechnologies (US2B – Nantes Université), ont analysé le génome de plusieurs de ces algues pour en comprendre l’évolution et l’impact sur les écosystèmes marins. Les résultats de cette étude ont récemment été publiés dans le Journal Cell.
EN SAVOIR PLUS :
https://u-news.univ-nantes.fr/biodiversite-le-genome-des-algues-brunes-mis-a-jour-par-des-chercheurs-nantais
Les membres de l’US2B s’associent à l’initiative française de défense de la science !
76 actualités/événements passés depuis 2015
L’orobanche du tournesol, Orobanche cumana, est la contrainte biotique la plus importante pour la production de graines de tournesol dans tous les pays où celui-ci est cultivé, sauf en Amérique du Nord et du Sud. Plusieurs stratégies de lutte ont été employées contre les orobanches, mais aucune d’entre elles n’a abouti à un succès sans équivoque. Actuellement, le moyen le plus efficace d’empêcher l’expansion des plantes parasites de racines est la résistance génétique, utilisée avec succès essentiellement chez le tournesol.
Le processus d’exploitation trophique par la plante parasite est régi par un dialogue moléculaire bien réglé entre les deux partenaires. Parmi les mécanismes sophistiqués qui permettent au parasite de détecter la présence d’une plante hôte et de coordonner son cycle de vie avec celle-ci, son processus particulier de germination est probablement le plus important. En effet, ce stade précoce du développement est essentiel à sa survie, car une graine germée qui ne parvient pas à se connecter à un hôte épuisera rapidement ses réserves d’énergie et mourra. Les orobanches compensent cela en ayant des exigences strictes pour la germination, impliquant la présence de molécules chimiques spécifiques exsudées par les racines de l’hôte, les stimulants de germination (GS).
On sait depuis des décennies que ces substances allélochimiques appartiennent principalement à la famille des strigolactones (SL), mais ce n’est que récemment que leur récepteur, une α/β-hydrolase (D14/KAI2), a été découvert. Cette interaction ligand/récepteur apparemment simple est en fait complexe. En effet, outre les SL, d’autres classes de métabolites secondaires végétaux ont été identifiées comme GS. Par exemple, le déhydrocostus lactone (DCL), une sesquiterpène lactone guaianolide (SqTL), est exsudé par les racines de tournesol et stimule spécifiquement la germination des graines d’O. cumana. Deuxièmement, la protéine réceptrice appartient à une famille multigénique KAI2 chez les plantes parasites de racines (au moins 8 séquences KAI2 chez O. cumana).
Le programme de recherche vise à identifier les récepteurs KAI2 des stimulants de germination (strigolactones et lactones sesquiterpéniques) chez la plante parasite O. cumana. Grâce à la disponibilité des séquences génomiques de plusieurs populations d’O. cumana, la biodiversité des récepteurs KAI2 sera évaluée et le polymorphisme observé sera ensuite corrélé à la réponse aux stimulants de germination dans les populations d’O. cumana grâce à un test à haut débit permettant d’évaluer la germination des graines de plantes parasites. Une sélection de séquences KAI2 représentatives de la diversité sera ensuite clonée pour la production des protéines correspondantes dans E. coli et la complémentation du mutant kai2-2 d’Arabidopsis. Les protéines recombinantes et les lignées d’Arabidopsis complémentées seront ensuite testées pour leur capacité et leur spécificité à lier et percevoir les stimulants de germination. Le travail doctoral proposé s’inscrit dans le cadre du programme ANR STIGO 2022-2025.
Dans de vastes zones des océans, l’azote biodisponible est rare. Certains micro-organismes tirent alors un avantage écologique de leur capacité à fixer une autre source azote, le diazote atmosphérique. Cette diazotrophie repose sur la nitrogénase, un complexe enzymatique coûteux en énergie et inhibé par l’oxygène. Si la diazotrophie a longtemps été attribuée aux cyanobactéries, des études récentes ont révélé l’existence d’une large diversité de diazotrophes non cyanobactériennes (DNCs) (Pseudomonadota, Planctomycetes, …). Dans ce contexte, Vibrio diazotrophicus a été utilisée comme modèle de DNCs marines. Le développement d’outils génétiques et d’un milieu expérimental adapté a permis de générer les différents mutants utilisés durant cette thèse, ainsi que de déterminer qu’une croissance en conditions micro-oxiques permettait l’activité diazotrophique chez cette souche. Cette stratégie repose sur une motilité (aérotactisme) et la formation de biofilms, permettant à la fois une respiration cellulaire et la protection de la nitrogénase. Chez cette DNC, la formation de biofilm et la libération d’azote impliquent l’activation d’un prophage conduisant à la lyse cellulaire. Une analyse transcriptomique comparative a révélé une forte versatilité métabolique lors de la croissance en N2, incluant la capacité d’utiliser de multiples sources azotées. NifA, RpoN et NtrC apparaissent comme des régulateurs clés dans l’expression et l’activité de la nitrogénase. De plus, l’expression de nitrogénase est restreinte à une sous-population, révélant une hétérogénéité phénotypique dans le processus de diazotrophie et suggérant une répartition des coûts énergétiques entre les cellules. Ces travaux mettent en lumière les mécanismes physiologiques et génétiques impliqués dans la diazotrophie. Ils ouvrent la voie à une meilleure compréhension du rôle des DNCs dans le cycle de l’azote marin.
L’US2B recrute un-e ingénieur-e biologiste dans le cadre d’un CDD de 18 mois.
Intitulé du poste : Production de lignées mutants perte-de-fonction par la technologie CRISPR-Cas9 et analyse des histones H2A chez la Diatomée
Type de poste : CDD
Durée du poste : 18 mois, renouvelable
Ville : Nantes
Nom du contact : Céline Duc
Email du contact : celine.duc@univ-nantes.fr
Laboratoire : CNRS UMR6286, US2B, Nantes Université
Plus de détails dont la Fiche de poste détaillée :
Actuellement, pour surveiller la malignité d’une tumeur, des biopsies sont nécessaires, mais cela représente un risque pour le patient car c’est invasif. De nouvelles techniques non invasives sont toujours en cours de développement afin d’assurer la surveillance en continue de la tumeur. Ainsi, les puces à protéines sont apparues comme essentielles dans le diagnostic, la découverte de médicaments et dans la protéomique. Dans ce contexte, on a développé un prototype d’imagerie multiparamétrique basé sur les avantages des cristaux photoniques qui est une alternative à la SPRi. Les données de cette étude ont démontré premièrement, qu’il était possible de détecter en temps réel et sans marquage, plusieurs interactions en simultanée. Dans un second temps, la technologie a été utilisée dans le suivi en temps réel du recrutement de la protéine RAD51, protéine centrale de la recombinaison homologue, sur l’ADNsb. Ainsi les résultats montrent une forte sensibilité de détection des évènements d’association-dissociation, permettant de calculer les paramètres cinétiques et ouvrent la voie pour un criblage à haut débit de nouveaux inhibiteurs de RAD51 impliqué dans la chimiorésistance. Dans ce même contexte, j’ai mené une étude dans la compréhension des mécanismes de régulation de RAD51, par la phosphorylation, ou encore par des cofacteurs tels que l’ATP et le calcium. Les résultats obtenus montrent une double régulation par le calcium, une fixation au niveau de la poche ATPase et pour la première fois, une fixation du calcium au niveau la région N-terminale de la protéine sur les résidus E16, E17 et E18.
En ce qui concerne la régulation de RAD51 par les MPTs, on a découvert une nouvelle phosphorylation sur la Ser97 du motif SRM. Ici on a démontré pour la première fois, que la PhosphoS97 de RAD51 augmente l’activité de la D-loop, la fixation et l’hydrolyse de l’ATP ainsi que l’affinité de RAD51 pour l’ADNsb.
Les bioconjugués sont de plus en plus utilisés dans les domaines des biotechnologies et de la médecine, offrant des opportunités pour le développement de vaccins en activant différentes voies immunitaires. Les vaccins glycoconjugués sont efficaces, bien tolérés et consistent en la conjugaison de polysaccharides de surface bactériens (PS) sur une protéine porteuse, permettant l’induction d’anticorps dirigés contre l’antigène glucidique grâce au recrutement de lymphocytes T CD4+.
Cependant, le processus actuel de production de ces glycoconjugués présente des limitations telles qu’une reproductibilité limitée, la génération de glycoconjugués mal définis menant à une antigénicité variable. En effet, la longueur du PS, le rapport protéine porteuse / PS et la chimie de conjugaison ont été établis comme des facteurs clés ayant un impact direct sur la réponse immunitaire.De plus, on suppose depuis longtemps que les sites auxquels le PS est attaché à la protéine porteuse (connectivité) peuvent également avoir un impact.
Ainsi, l’importance du caractère homogène du glycoconjugué sur la réponse immunitaire peut maintenant être abordée grâce à une combinaison d’outils moléculaires telle que la mutagenèse sélective, l’incorporation d’acides aminés non canonique (ncAA) et des stratégies de conjugaison bio-orthogonales.
Cette thèse vise également à évaluer l’efficacité de la co-délivrance des composants du vaccin en réalisant une liaison covalente entre un adjuvant et le glycoconjugué. Notre modèle d’étude ciblant Streptococcus pneumoniae (Pn), un polysaccharide synthétique mimant la capsule du sérotype 14 de Pn (Pn14PS) est utilisé comme antigène glycanique.
Deux approches sont proposées : la première utilise une protéine hautement conservée appelée adhésine de surface pneumococcique A (PsaA), tandis que la seconde utilise la flagelline dérivée de Salmonella enterica serovar Typhimurium (FliC) qui agit à la fois comme protéine porteuse et adjuvant en activant la signalisation du récepteur Toll-like 5 (TLR5). Profitant de l’absence de cystéine dans PsaA et FliC, jusqu’à quatre résidus ont été mutés en cystéines pour être ensuite conjugués à Pn14PS en utilisant la chimie de couplage thiol-maléimide. Les sites de conjugaison sont ciblés de manière stratégique pour étudier l’impact de la connectivité sur la réponse humorale induite et pour éviter toute interférence antigénique avec les épitopes B propres à S. enterica.
L’incorporation d’un ncAA a été étudiée afin de lier le glycoconjugué élaboré à partir de PsaA à un adjuvant, permettant ainsi d’évaluer l’impact du couplage covalent entre le vaccin et l’adjuvant.
La radiothérapie à Ultra-Haut Débit de Dose (RT-UHDD), de l’ordre du Gray/msec, est une technique prometteuse pour réduire la toxicité aux tissus sains. Par ailleurs, la balistique des hadrons (protons/ions d’hélium) permet un meilleur ciblage des tumeurs, limitant la dose délivrée aux tissus sains environnants. Nous avons émis l’hypothèse que la hadronthérapie à UHDD est la méthode optimale pour limiter les séquelles.
Nos travaux montrent que la hadronthérapie UHDD préserve le développement d’embryons de poisson-zèbres par rapport à la radiothérapie conventionnelle (conv-RT). Par expression transcriptionnelle, nous avons montré que l’arrêt du cycle cellulaire est moins affecté après la protonthérapie UHDD (PT-UHDD) qu’après la conv-PT. Également, la PT-UHDD a montré moins de dommages à l’ADN et d’apoptose. Nous avons ensuite étudié les effets précoces de la PT-UHDD sur le système vasculaire, en utilisant des embryons de poisson-zèbres transgéniques. Nos résultats montrent que les vaisseaux existants ne sont pas structurellement affectés par l’irradiation. Par contre, la formation de nouveaux vaisseaux est bloquée. Des résultats similaires ont été obtenus avec des pseudo-tubes formés par des cellules endothéliales in vitro. Cependant, une augmentation des péricytes autour des vaisseaux inter-segmentaux a été observée après PT-UHDD. De plus, par RT-qPCR, nous avons observé une augmentation de gènes de réponse vasculaire après PT-UHDD, notamment la VE-cadhérine.
En conclusion, ce travail met en évidence une protection du développement des embryons de poisson-zèbre par la hadronthérapie UHDD. Ce phénotype semble partiellement indépendant d’une meilleure protection vasculaire.
Le but du GDR EPIPLANT est de regrouper la communauté scientifique qui étudie les mécanismes épigénétiques contribuant au développent et aux réponses adaptives chez les organismes photosynthétiques.
L’équipe « Epigénomique des Microalgues et interactions avec l’environnement » fait partie des équipes participantes.
Suite des portaits sur « les Femmes au Cœur de la Science à l’US2B », nous vous invitons à découvrir Pauline CRETIN, doctorante en troisième année dans l’équipe « Ingénierie Moléculaire et Glycobiologie » au sein du laboratoire.
Suite des portaits sur « les Femmes au Cœur de la Science à l’US2B », nous vous invitons à découvrir Isadora Panez, doctorante en troisième année dans l’équipe « Mécanisme et régulation de la réparation de l’ADN » au sein du laboratoire.
Suite des portaits sur « les Femmes au Cœur de la Science à l’US2B », nous vous invitons à découvrir Gaelle SAADE, doctorante en troisième année dans l’équipe « Mécanisme et régulation de la réparation de l’ADN » au sein du laboratoire.
L’objectif principal de la thèse est d’étudier les rôles des acteurs non chromatiniens dans le maintien de l’épigénome au cours du cycle cellulaire en caractérisant l’influence de la perte de l’enzyme E(z) chez la diatomée, Phaeodactylum tricornutum. Cette espèce possède un génome séquencé. De plus, elle présente différents morphotypes (ovale, fusiforme, triradié, cruciforme). Son épigénome a été caractérisé pour de nombreuses marques épigénétiques comme H3K27me3. L’enzyme E(z) est conservée chez cette espèce et elle est nommée PtEZH. En outre, cette diatomée présente un cycle cellulaire bien caractérisé qui dure environ 10h, et il est possible de synchroniser en phase G1 les cellules de P. tricornutum grâce à une incubation prolongée à l’obscurité. Afin d’étudier les rôles d’E(z) sur le maintien de la marque épigénétique H3K27me3 au cours du cycle cellulaire chez P. tricornutum, des lignées perte-de-fonction pour l’enzyme PtEZH seront produites par conjugaison bactérienne avec la méthode CRISPR-Cas9 dans les 4 morphotypes de Phaeodactylum tricornutum. Les effets de la perte de l’activité de PtEZH seront évalués sur l’abondance et la localisation de la marque H3K27me3 à l’échelle nucléosomale au cours du cycle cellulaire.
Nous recherchons un étudiant de master 2 pour un stage de 6 mois. Le projet proposé vise à étudier le maintien de l’épigénome via l’étude des histones H3 et des enzymes impliquées dans le dépôt et le retrait des modifications post-traductionnelles portées par les histones H3. Le modèle d’étude est la diatomée Phaeodactylum tricornutum, une algue marine unicellulaire. L’étudiant mettra en œuvre des approches de biologie moléculaire et de biochimie (RT-PCR quantitative, western-blot, CRISPR-Cas9, comptage cellulaire et/ou cytométrie de flux) ainsi que de la bioinformatique (blast, modélisation moléculaire).
Le stage sera co-encadré par Dr-HDR C. Duc pour l’aspect expérimental et Dr-HDR S. Téletchéa pour les analyses bioinformatiques
Contact: Céline Duc, Maître de conférences HDR, celine.duc@univ-nantes.fr
L’objectif du GDR EPIPLANT est de fédérer la communauté scientifique française qui étudie les mécanismes épigénétiques contribuant au développement et aux réponses adaptatives chez les organismes photosynthétiques terrestres et marins.
En réponse aux fluctuations environnementales, les organismes unicellulaires et multicellulaires sont capables d’exprimer différents phénotypes à partir d’un génotype donné, en employant de multiples mécanismes qui impliquent des processus épigénétiques.
L’équipe « Épigénomique des Microalgues et Interactions avec l’environnement » a rejoint le GDR EPIPLANT et Céline Duc participe au comité scientifique dirigé par Aline Probst.
Plus d’infos: https://gdr-epiplant.preprod.lamp.cnrs.fr/
Les cassures double brin de l’ADN sont les plus délétères pour une cellule. L’une des voies de réparation de ces dommages est la recombinaison homologue (RH) impliquant la protéine RAD51. Cette recombinase joue un rôle central dans la RH et est surexprimée dans les cellules cancéreuses. La réparation accrue des dommages à l’ADN par les cellules tumorales conduit le plus souvent au développement de résistances aux thérapies anticancéreuses, limitant leur efficacité. RAD51 a déjà fait l’objet de ciblage par différentes approches. Son inhibition a notamment permis de sensibiliser les cellules cancéreuses aux traitements. Nous avons ici pu mettre en évidence des petites molécules capables de moduler l’activité de RAD51 in vitro et également de sensibiliser des cellules cancéreuses en association avec le cisplatine.
Dans le cadre d’un projet financé par le Fonds de Développement de la Pédagogie 2022-2023 de la Faculté des sciences et des techniques, une équipe regroupant des enseignants (Audey Queudet et Stéphane Téletchéa) et des étudiants en biologie a produit un cycle de vidéos autour de la thématique de la bioinformatique. Ces ressources pédagogiques audiovisuelles, conçues par les étudiants et à destination des étudiants, sont publiées sur la WebTV de Nantes Université.
L’épigénétique est un domaine en pleine expansion au sein de la science contemporaine, qui se définit par étant les modifications héritables de la régulation génique sans altération de la séquence de l’ADN. Parmi les microalgues, la diatomée marine Phaeodactylum tricornutum s’est révélée être un organisme modèle de choix pour l’étude de l’épigénétique. Cette thèse a pour objectif d’approfondir notre compréhension du paysage épigénétique de P. tricornutum en utilisant la dernière version assemblée de son génome, afin de cartographier et caractériser l’épigénome. Afin de faciliter l’analyse des données épigénomiques et des transcrits, nous avons développé un navigateur exhaustif appelé PhaeoEpiView (https://PhaeoEpiView.univ-nantes.fr). Dans un souci de précision et d’exhaustivité accrues, nous avons mis à jour les marques d’histones précédemment publiées en recourant à des anticorps monoclonaux plutôt que polyclonaux, et nous avons utilisé une profondeur de séquençage plus importante. Par ailleurs, PhaeoEpiView a été enrichi en intégrant de nouvelles marques épigénétiques, H3K27Ac et H3K36me3, qui jouent un rôle crucial dans l’activation transcriptionnelle chez P. tricornutum. Parallèlement, nous avons exploré la fonctionnalité des protéines du complexe Polycomb (PcG), en particulier PRC1 et PRC2, qui sont impliquées dans la répression globale des gènes. Nos travaux ont porté sur l’étude de ces protéines chez P. tricornutum, tout en examinant les éventuelles différences par rapport aux organismes multicellulaires. Une découverte majeure a été réalisée grâce à la mise en évidence d’un modèle de cooccurrence unique de marques épigénétiques chez les diatomées, suggérant un mécanisme coopératif de répression et un recrutement interdépendant. En parallèle, afin d’approfondir notre compréhension du succès écologique de cette diatomée, nous avons étudié l’interaction entre le microbiome et l’épigénétique dans la réponse de P. tricornutum aux stress environnementaux, notamment l’hypersalinité. Nos recherches ont démontré la dépendance de P. tricornutum à l’égard de son microbiome associé pour sa survie dans des environnements à forte salinité. De plus, nous avons examiné l’influence de la méthylation de l’ADN sur la plasticité phénotypique de cet organisme exposé l’hypersalinité. Dans l’ensemble, nos résultats présentent des implications majeures pour le domaine de l’épigénétique, en mettant en évidence les mécanismes moléculaires régissant l’expression génique et les réponses environnementales chez les microalgues, et au-delà.
Etude de la régiosélectivité de la glucosylation enzymatique de polyphénols à l’aide de mutants des sucrose phosphorylases de Bifidobacterium adolescentis et d’Alteromonas mediterranea. Pour ce faire, quatre polyphénols ont été considérés : le resvératrol (stilbène), la (+)-catéchine (flavan-3-ol), la quercétine et le kaempférol (flavonols).
Les enzymes natives et leurs mutants ont été produits, purifiés et caractérisés. Leur capacité à catalyser la transglucosylation a été évaluée sur les quatre polyphénols cibles. Les réactions enzymatiques ont été suivies par HPLC analytique et les produits ⍺-glucosylés séparés par HPLC préparative. Des analyses par UPLC-MS, RMN et spectrométrie de masse ont permis de valider les structures des composés ⍺-glucosylés obtenus. Les interactions stabilisantes entre les polyphénols et le site actif des enzymes ont été déterminées par une étude in silico d’arrimage moléculaire.
La combinaison de l’approche expérimentale de synthèse et d’identification des formes glucosylées des polyphénols, et de l’approche de modélisation bioinformatique par ancrage moléculaire a permis d’apporter des éléments nouveaux de compréhension du mécanisme de glucosylation des sucrose phosphorylases.
Les conclusions de ces recherches pourront mener au développement de nouveaux outils enzymatiques afin de contrôler à façon la régiosélectivité de la glucosylation des polyphénols par des sucrose phosphorylases.
Titre: Conserving the alien plant: the biology and the story of Rafflesiaceae.
Epigenetics (heritable changes, through mitotic or meiotic cell division, in gene expression without modifications of the underlying DNA sequence) has emerged as an important discipline bridging environment, phenotypic plasticity and evolution. Being dynamic and reversible, epigenetic-induced variations allow a greater phenotypic plasticity in response to a changing environment, and may therefore facilitate acclimation and ultimately adaptation upon its action on DNA sequence. Epigenetic mechanisms have been extensively investigated in plant and animal genomes, yielding insightful findings into genome regulation and impacts on cell homeostasis, development, diseases and even evolution, in particular from population epigenomics studies. Despite the growing interest in epigenetics and the development of sequencing technologies, knowledge on these processes remains scarce in species outside animals and plants, in particular microbial eukaryotes such as stramenopiles, rhizaria, alveolates and ciliates. Considering the position of these species in the tree of life, investigating their epigenome will help to understand the evolutionary origins of epigenomic components and mechanisms as well as their role in evolution and speciation.
This Special Issue welcomes original research, review, and methodology papers on the epigenetics of model and non-model organisms from protist lineages using multidisciplinary approaches and/or high-throughput technologies to investigate processes such as cell differentiation, sexual reproduction, development, stress response, acclimation and adaptation to the environment.
Dr. Leila Tirichine
Dr. Susana Coelho
Guest Editors
L’augmentation du parasitisme de l’orobanche rameuse, plante parasite racinaire obligatoire du colza d’hiver dans l’ouest français, couplée à sa diminution localisée sur quelques parcelles au sein du même contexte pédoclimatique, suggèrent que l’interaction entre les deux plantes est multifactorielle et doit être mieux appréhendée. A partir de l’étude de sols présentant deux niveaux d’infestations, dits compatibles et suppressifs, ces travaux soutiennent l’hypothèse d’une interaction tripartite avec le microbiote du sol. Dans un premier temps, ce dernier contribuerait à amplifier le signal moléculaire rhizosphérique, nécessaire pour la reconnaissance du colza par l’orobanche. Plus précisément, des activités enzymatiques spécifiques au métabolome du colza (myrosinases) permettant l’induction de la germination de l’orobanche, ainsi que la production de molécules signales initiant la formation du parasitisme, ont été détectées et fonctionnellement validées chez des bactéries et champignons des deux types de sol. Dans un second temps, le suivi de l’évolution du microbiote du sol et de la rhizosphère durant le parasitisme a permis de valider les observations de terrains sur les sols suppressifs et surtout de mettre en évidence des microorganismes ciblant spécifiquement l’orobanche. Plus précisément, certaines associations microbiennes ont été positivement corrélées à la réduction de l’infestation et à l’apparition de symptômes nécrotiques. Finalement, deux candidats fongiques ont été proposés à partir de la mise au point d’une méthode de détection de nécroses par analyse d’image. Cette étude a permis d’approfondir la communication allélopathique entre les plantes hôtes, les plantes parasites et leurs microbiotes associés.
The Special Edition “Plant Chromatin Biology” will focus on the molecular players that operate within the chromatin structure and composition and how they can be regulated in response to environmental cues or developmental transitions. With their strong plasticity, plants offer great opportunities to decipher the mechanisms that operate within chromatin and to understand how the chromatin structure and the epigenetic information ensure the fine tuning of the genome expression program in various conditions.

L’ostéosarcome est une tumeur osseuse primaire rare, qui touche principalement les enfants et les adolescents. C’est une maladie de mauvais pronostic, en particulier pour les patients atteints d’une maladie métastatique. Une mauvaise réponse thérapeutique aux traitements conventionnels comme la chimiothérapie est observée avec le développement de métastases pulmonaires, soulignant la nécessité d’améliorer les schémas thérapeutiques actuels et l’identification de marqueurs précoces de la maladie récurrente et métastatique. Les cellules tumorales circulantes (CTC) jouent un rôle clé dans le processus métastatique et pourraient être de puissants biomarqueurs de la maladie évolutive. La présente étude visait à isoler les CTCs en utilisant un modèle préclinique d’ostéosarcome humain et de suivre leur cinétique de libération et leur modulation par l’ifosfamide. Les CTCs étaient détectables dans circulation sanguine avant toute tumeur primaire palpable. L’ifosfamide a augmenté le nombre de CTC et, au contraire, a diminué le nombre de nodules tumoraux pulmonaires. Sur les tumeurs établies, l’ifosfamide a ralenti la croissance tumorale et n’a pas moduler le nombre de CTC qui pourrait s’expliquer par une libération de cellules cancéreuses de la tumeur primaire avec une réduction des propriétés d’induction de métastases pulmonaires. Cette étude met en évidence l’intérêt biologique des CTCs dans l’ostéosarcome comme marqueur précoce de la maladie récidivante.
Titre: Le resvératrol et ses formes glycosylées comme alternatives thérapeutiques pour le traitement de la maladie de Parkinson.
La maladie de Parkinson est une maladie neurodégénérative progressive jusqu’à présent incurable. Le traitement de la maladie est uniquement symptomatique et repose sur le remplacement de la dopamine et sur la mise en œuvre d’approches non dopaminergiques. Bien que ces traitements améliorent la fonction motrice ils déclenchent également des effets indésirables problématiques. Il est donc important de rechercher des molécules pouvant agir dans la prévention ou le traitement de la maladie de Parkinson.
Dans ce contexte, un polyphénol, le resvératrol, a été mis en évidence pour ses activités anti-inflammatoires et antioxydantes. L’optimisation structurale par glycosylation de ce composé apparaît comme une alternative thérapeutique.
Titre: Evolution of Strigolactone perception in land plants.
Alexandre de Saint Germain obtained his PhD thesis in 2012 at IJPB-INRAE (France) in the group of C Rameau, where he studied Strigolactone signalling. After a first post doc at the SALK Institute (La Jolla, USA) working on the Strigolactone receptor family (2013-2016) and a second Post doc at IJPB (2016-2020) on allelochemicals perception, he obtained, in 2020, a permanent position at INRAE. He is now developing a new thematic on allelopathy in the Strigolactone and Allelochemical Signalling (SAS) Team at IJPB. His current research aims to decipher the genetic and molecular mechanisms of chemical belowground plant-plant interactions.
Titre: Quand la science a du nez : la perception des odeurs vue au niveau atomique.
Les mammifères, pour évoluer dans leur environnement, font constamment appel à leurs cinq sens : la vue, l’ouïe, le toucher, le goût et l’odorat. Si tout le monde sait nommer la perte de la vue ou de l’ouïe, très peu connaissaient le terme lié à la perte de l’olfaction avant la pandémie de COVID-19 : l’anosmie. C’est très révélateur du peu de connaissances que nous avons sur le fonctionnement de notre odorat. La perception des odeurs est basée sur les récepteurs olfactifs (RO), qui appartiennent à la grande famille des récepteurs couplés aux protéines G. Les gènes codant pour ces protéines n’ont été découverts chez le mammifère qu’en 1991 par Linda Buck et Richard Axel (prix Nobel de médecine 2004), ce qui signifie que l’homme a fait ses premiers pas sur la lune avant de savoir comment il sentait son café. Depuis, des progrès rapides ont été réalisés par la communauté scientifique pour mieux comprendre les mécanismes impliqués dans la perception des odeurs. Ici, je présenterai les liens établis aujourd’hui entre la perception d’une odeur et l’émotion qu’elle déclenche jusqu’aux engrenages moléculaires rendant les RO sensible aux molécules odorantes. Ce progrès est possible grâce à une combinaison d’expertises mêlant analyse sensorielle, biologie cellulaire, biochimie et analyse structurale. J’ouvrirai également la discussion sur les possibilités que l’étude de l’olfaction pourrait ouvrir et leur impact sur notre vie quotidienne.
Titre: Boucles de rétroaction éco-évolutives et génomiques chez les plantes parasites.
Chez les plantes, la transition d’un mode de vie libre au parasitisme (spoliation de l’eau et des nutriments d’une autre plante) offre un avantage éco-évolutif important dans des environnements hautement compétitifs et pauvres en ressources. La transition des plantes vers un mode de vie non photosynthétique entraîne des changements morphologiques extrêmes et des reconfigurations génétiques massives. Cet exposé explore la progression du syndrome de réduction parasitaire au niveau génétique et explique comment le parasitisme affecte les modèles de diversification. La diversité naturelle des spécialisations parasitaires et des tests d’hypothèses phylostatiques révèlent que l’établissement d’un parasitisme obligatoire déclenche des reconfigurations génomiques. Après la perte éventuelle de la photosynthèse, les modifications fonctionnelles-génomiques progressent rapidement, accompagnées d’une accélération progressive des taux d’évolution moléculaire. La transition vers un parasitisme obligatoire déclenche apparemment une boucle de rétroaction éco-évolutive qui affecte tous les compartiments génomiques, ce qui pourrait favoriser la spécialisation trophique. Dans ce cas, les modèles de diversification changent « en route vers » l’holoparasitisme, ce qui nous amène à supposer que la diversification des parasites est co-façonnée par une dépendance croissante envers leurs plantes hôtes, ce qui crée une barrière isolante absente des plantes ordinaires.
Titre: Protein local conformations analyses in ordered and intrinsically disordered proteins in the light of a structural alphabet.
Protein structures are highly dynamic macromolecules. Molecular dynamics (MDs) simulations were performed on 169 representative protein domains. Classical secondary structures were explored. Concerning the helical structures, only 76.4% of the residues associated to α-helices retain the conformation; this tendency dropped to 40% for 310- and for π-helices [1]. The rigidity of β-sheet was confirmed, but showed its capacity to transform into turns. Finally, turns converted easily to helical structures while bends prefer the extended conformations. Protein Blocks structural alphabet [2] showed that the majority of PBs remains with high frequency in original conformation. Few PBs have a higher tendency to be more flexible. The intriguing fact was that the change from a PB to another one did not correspond to a simple geometrical evolution. It was more frequent to go to an unexpected PB than an expected one [3]. Disorder protein ensembles were analysed with PBs allowing to quantify the continuum from rigidity to flexibility and finally disorder [4,5]. These results have been compared to different types of prediction [6,7].
1. Narwani, T.J.; Craveur, P.; Shinada, N.K.; Santuz, H.; Rebehmed, J.; Etchebest, C.; de Brevern, A.G. Dynamics and deformability of α-, 310- and π-helices. Archives of Biological Sciences 2018, 70, 21-31.
2. de Brevern, A.G.; Etchebest, C.; Hazout, S. Bayesian probabilistic approach for predicting backbone structures in terms of protein blocks. Proteins 2000, 41, 271-287.
3. Narwani, T.J.; Craveur, P.; Shinada, N.K.; Floch, A.; Santuz, H.; Vattekatte, A.M.; Srinivasan, N.;
Rebehmed, J.; Gelly, J.C.; Etchebest, C., et al. Discrete analyses of protein dynamics. J Biomol Struct Dyn 2019, 1-15.
4. Melarkode Vattekatte, A.; Narwani, T.J.; Floch, A.; Maljkovic, M.; Bisoo, S.; Shinada, N.K.; Kranjc, A.;
Gelly, J.C.; Srinivasan, N.; Mitic, N., et al. Data set of intrinsically disordered proteins analysed at a local protein conformation level. Data Brief 2020, 29, 105383.
5. Melarkode Vattekatte, A.; Narwani, T.J.; Floch, A.; Maljkovic, M.; Bisoo, S.; Shinada, N.K.; Kranjc, A.;
Gelly, J.C.; Srinivasan, N.; Mitic, N., et al. A structural entropy index to analyse local conformations in intrinsically disordered proteins. J Struct Biol 2020, 210, 107464.
6. de Brevern, A.G. Analysis of protein disorder predictions in the light of a protein structural alphabet. Biomolecules 2020, 10.
7. de Brevern, A.G. Impact of protein dynamics on secondary structure prediction. Biochimie 2020, 179, 14-22.
L’UFIP recrute un-e ingénieur-e d’études en bioinformatique dans le cadre d’un CDD de 12 mois renouvelable.
Intitulé du poste : Analyse de données haut débit de type RNA Seq, Meta transcriptomics et Meta Genomics
Type de poste : CDD
Durée du poste : 12 mois, renouvelable
Ville : Nantes
Nom du contact : Leila Tirichine Delacour
Email du contact : tirichine-l@univ-nantes.fr
Laboratoire : CNRS UMR6286, UFIP, université de Nantes
Description du poste:
Un poste d’ingénieur d’études en bioinformatique est ouvert dans l’équipe «Epigénomique des microalgues et interactions avec l’environnement dans l’UMR CNRS 6286, à l’université de Nantes afin de travailler sur des données de type RNA seq (transcriptome), meta transcriptomique, meta génomique. Le travail portera sur l’analyse à large échelle des données de séquençage obtenues dans l’équipe sur des modèles de micro-algues et des communautés bactériennes.
Ce poste est un CDD de 12 mois renouvelable
Le (/la) candidat(e) doit être soit un(e) biologiste ayant une bonne maîtrise et une expérience de la bioinformatique, soit un(e) bioinformaticien(ne) ayant de solides connaissances en biologie. Le (/la) candidat(e) doit avoir une première expérience dans l’analyse de données NGS (en particulier de type metagénomique), avoir de fortes compétences en programmation (en particulier en Python ou en R) et être familier avec les environnements de calcul parallèle (en particulier SGE ou SLURM). Il (/elle) doit également montrer un intérêt pour le développement de nouvelles approches. De bonnes connaissances en statistiques serait un plus.
Le(/la) candidat(e) doit être motivé(e) par les questions biologiques et disposé(e) à interagir avec les différents membres de l’équipe. De bonnes qualités relationnelles et de communication sont essentielles afin que le(/la) candidat(e) puisse échanger et partager ses connaissances dans un environnement essentiellement composé de biologistes.
Votre candidature inclura une lettre de motivation, un CV et le nom d’au moins deux référents et doit être envoyée à Leila Tirichine (tirichine-l@univ-nantes.fr)
L’UFIP et le département de biologie de la Faculté des Sciences et Techniques recrutent *un.e technicien.ne biologiste* dans le cadre d’un CDD de 12 mois.
La personne recrutée devra réaliser des missions de recherche et d’enseignement. Elle devra mettre en œuvre, dans le cadre de protocoles établis les techniques de biologie cellulaire et moléculaire et de biochimie pour la préparation, la caractérisation et l’étude d’échantillons biologiques. Assurer l’entretien de cultures biologiques.
Les activités de recherche s’effectueront au sein de l’équipe « Epigénomique des microalgues et interactions avec l’environnement » afin d’apporter un soutien technique.
Dans le cadre des activités d’enseignement, la personne recrutée participera à la préparation de travaux pratiques réalisés au sein du département de biologie, en biologie moléculaire, microbiologie et biologie cellulaire, ainsi qu’à l’entretien, au fonctionnement et à la petite maintenance des appareils utilisés lors des travaux pratiques.
Le temps consacré aux activités d’enseignement et de recherche sera respectivement de 50%.
Pour plus de détails, consulter l’offre via le lien ci-après :
https://www.univ-nantes.fr/travailler-a-luniversite/offres-demploi-et-concours/luniversite-de-nantes-recrute-un-e-technicien-ne-biologiste-pour-la-faculte-des-sciences-et-des-techniques-4
Date limite de réception des candidatures : 23/09/2021
Titre: Quality Control in Protein synthesis: when bacteria cope with ribosome stalling.
Bacteria cope with ribosome stalling thanks to trans-translation, a major quality control system of protein synthesis that is mediated by tmRNA, an hybrid RNA with properties of both a tRNA and an mRNA, and the small protein SmpB. Because trans-translation is absent in eukaryotes but necessary for bacterial fitness or survival, it is a promising target for the development of novel antibiotics. Here I will present four cryo-EM structures of the ribosome during trans-translation. These include the first high-resolution structure of the whole pre-accommodated state, as well as structures of the accommodated state, the translocated state, and a new translocation intermediate. Together, they shed light on the movements of the tmRNA-SmpB complex in the ribosome, from its delivery by the elongation factor EF-Tu to its passage through the ribosomal A and P sites after the opening of the ribosomal bridges. I will also present a medicinal chemistry program that we perform to discover new and specific compounds targeting trans-translation in resistant bacteria.
La crise sanitaire liée à l’émergence du virus SARS-CoV-2 a propulsé la vaccination et sa nécessité sur le devant de la scène. Le colloque « Vaincre la Covid-19 par la vaccination et l’immunothérapie », qui a eu lieu dans le cadre des Journées Scientifiques de l’Université de Nantes, le 1er juin, a été propice à décrire l’état de l’art et l’implication des laboratoires et des entreprises ligériennes dans la lutte anti-COVID19.
| Anne-Marie Moulin | L’évolution de l’immunisation, de la protection des enfants à l’immunisation du corps de la nation |
| Bruno Pitard | Vaccination à ADN ou à ARN contre le SARS-CoV-2 |
| Nicolas Poirier | Vaccination multi-épitopes Lymphocytes T contre-stratégie de protection large contre les variants du SARS-CoV-2V |
| Bernard Vanhove | Développement d’anticorps polyclonaux anti-SARS-CoV-2 |
| Jacques Le Pendu | Associations Covid-19 et groupes sanguins ABO, quels liens avec l’immunité ? |
| Patrick Peretti-Watel | Hésitation vaccinale et Covid-19 ? |
Titre: Kinetic target-guided synthesis et acides hydroxamiques des outils efficaces pour la drug-discovery ciblant les metalloproteases.

Une conférence PIRAMID sera donnée par Rebecca DEPREZ-POULAIN le 26 mars 2021, à 14 heures, en visio.
Lien zoom pour assister à la conférence : https://univ-nantes-fr.zoom.us/j/98841532048
Dans le cadre de la thèse de Lydia Ogonda (doctorat conjoint avec l’Université de Nairobi), de nouvelles cellulases alcaliphiles de la biodiversité kényane ont été identifiées et améliorées pour leur efficacité sur la cellulose cristalline grâce au génie moléculaire.
Sous la direction du professeur Charles Tellier de l’UFIP en collaboration avec le professeur Edward K.Muge et le professeur Francis J.Mulaa de l’Université de Nairobi, avec l’aide d’Amélie Saumonneau de D-ZYME, Lydia Ogonda a identifié, cloné et caractérisé biochimiquement deux nouvelles cellulases, BpGH9 et BpGH48, à partir d’une souche de Bacillus pumilus isolée du Lac Bogoria au Kenya. Ils ont découvert que la BpGH9 est une endocellulase modulaire appartenant à la famille des glycosides hydrolases 9 (GH9), qui contient un module catalytique (GH) et un module de liaison aux glucides appartenant à la classe 3 et à la sous-classe c (CBM3c). Fait intéressant, ils ont trouvé que cette enzyme était extrêmement tolérante à un pH alcalin élevé et reste significativement active à pH 10. Quant à BpGH48, il s’agit d’une exocellulase, appartenant à la famille des glycosides hydrolases 48 (GH48) et agit sur l’extrémité réductrice de l’oligo -β1,4 glucanes. Ils ont construit une forme tronquée de BpGH9 et une fusion chimérique avec un module CBM3a supplémentaire. Ils ont constaté que la suppression du module CBM3c entraîne une baisse significative de l’activité catalytique. Ils ont également découvert que la fusion de CBM3a, bien que dans une position non native, augmentait l’activité de BpGH9 sur la cellulose cristalline.
Pour plus de détails, consultez l’article paru dans Biotechnology Letters (Springer Ed).
La plateforme IMPACT de l’UFIP a contribué à une étude qui vient d’être mise en ligne sur BioRxiv. Fruit d’une collaboration entre Cathy Charlier, Pierre Weigel et l’équipe de Vincent Parissi du laboratoire de Microbiologie Fondamentale et Pathogénicité (MFP) UMR 5234 CNRS-Université de Bordeaux, cette étude caractérise davantage l’interaction entre la protéine virale Spike du Sars-Cov2 et le récepteur ACE2 et les processus cellulaires engagés dans l’étape d’entrée du virus. Cette collaboration a mis en place diverses approches in silico, in vitro et in cellulo qui nous ont permis de suivre spécifiquement l’association Spike / ACE2. La plateforme IMPACT a en particulier participé à la caractérisation de cette association par des approches de biophysique disponible à l’UFIP comme la technique AlphaLISA et le biolayer interferometry (BLI).
Pour en savoir plus :

Au 1er février, le site Internet de l’UFIP fait peau neuve. Bonne visite !


Financé par la Région Pays de La Loire, le but du projet SMIDIDI porté par François Delavat, jeune maître de conférences recruté en 2019 à l’UFIP au sein de l’équipe « Epigénomiques des microalgues en lien avec l’environnement » dirigée par Leila Tirichine, est d’amorcer des connaissances sur le comportement social des bactéries, par le développement d’un modèle d’étude composé d’une souche bactérienne vivant en association symbiotique avec un micro-eucaryote, une micro-algue.
François Delavat souhaite déverrouiller les connaissances encore fragmentaires sur le comportement des bactéries vivant en contact étroit avec des micro-algues photosynthétiques marines. Ce micro-environnement immédiat, appelé phycosphère, est le lieu d’échanges moléculaires entre la micro-algue et son cortège bactérien associé. Les bactéries présentes au sein de cette phycosphère sont donc soumises à des conditions environnementales différentes du milieu environnant (la colonne d’eau).
Les résultats de ce projet, en lien avec la philosophie du programme ERC, peuvent potentiellement aboutir à un changement de paradigme dans l’étude des interactions bactéries – micro-eucaryote.
Le dispositif régional « Etoiles Montantes » vise à soutenir les chercheurs et enseignants-chercheurs les plus « prometteurs » attestant d’un haut niveau scientifique et d’un potentiel permettant d’assurer à court terme le dépôt d’une candidature à l’ERC Starting Grant ou Consolidator Grant.
Leila Tirichine Delacour, Directrice de Recherche CNRS à l’UFIP co-porte le projet 100 Diatom Genomes Project avec Thomas Mock de l’Université East Anglia (UK). Ce projet, d’une durée de 3 ans, implique 32 laboratoires partenaires et est financé par le Joint Genome Institute (USA).
L’UFIP comptera dans ses effectifs à partir du 1er février 2021 Dr Franck Bertorelle, un ingénieur de recherche dans le domaine des nanoparticules. Il développera en lien avec les travaux de Cyrille Grandjean sur la conception de nouveaux vaccins et avec Fabrice Fleury sur la conception de nouveaux outils de détection biotechnologiques.
Franck Bertorelle a une formation de chimie supramoléculaire et a précédemment été en poste à l’Institut Lumière Matière de Lyon.
Diatoms are one of the ecologically most successful eukaryotic phytoplankton in the world. They are abundant in a wide range of habitats, their physiology, plasticity and fast adaptation to different environmental conditions help them dominate modern Oceans. Compared to genetic regulation, epigenetic changes can be flexible and reversible and histone modifications are one of the epigenetic mechanisms which can impact gene expression. Phaeodactylum tricornutum (P. tricornutum) is one of the model diatom species, also the first unicellular organism with a full repertoire of post-translational modifications of histones, which makes it an ideal species to study epigenetic regulation in single celled organisms. In this thesis manuscript, I focus on histone modification mechanisms in P. tricornutum, utilize classical reverse genetic method: knockout of candidate genes to identify the catalytic enzyme which is responsible of the deposition of histone modifications. Polycomb group protein (PcG) complexes are evolutionarily conserved epigenetic regulatory components that act antagonistically with Trithorax (TrxG) complexes to regulate genes which are involved in cell differentiation and development. In the first chapter we investigated the diversity of PcG and TrxG genes in marine unicellular species, report the correlation of these epigenetic modifiers and environmental factor for the first time, also emphasise the unique co-occurrence pattern of histone marks in P. tricornutum. Based on those discoveries, further study with chapter two and three focused on two PcG complexes, PRC2 and PRC1. In total, three core components of PcG protein were identified in PRC1 and PRC2 complex respectively, the second part of thesis explored the unique function of PRC2 and its associated mark H3K27me3 which I report related to morphology in P. tricornutum. Chapter three discussed the crosstalk between H3K27me3 and H2AK119Ubi which is deposited by PRC1. The last chapter describes a novel histone modification detected in P. tricornutum was found conserved among eukaryotes. The last chapter reports the characterization of this novel mark and identification of the histone writer.
La réparation des dommages de l’ADN durant les thérapies anticancéreuses peuvent contribuer à la résistance des cellules tumorales, limitant ainsi l’efficacité du traitement. Chez l’homme, la réparation des cassures double-brin (CDB) de l’ADN par recombinaison homologue (RH) fait intervenir la protéine Rad51.
Cette protéine s’est révélée être impliquée dans les problèmes de résistance aux traitements anticancéreux puisque son inhibition par différentes approches a pour effet de sensibiliser les cellules cancéreuses aux traitements. Rad51 constitue donc une cible intéressante. Dans ce contexte la recherche d’inhibiteurs de Rad51 permet de proposer des solutions thérapeutiques afin d’améliorer les traitements anticancéreux actuels. Nous avons montré que des petites molécules étaient capables de moduler spécifiquement l’activité de Rad51 _in vitro_ et nos résultats cellulaires ont montré un effet chimiosensibilisateur.
Les vaccins glycoconjugués qui permettent d’obtenir une réponse protectrice dirigée contre les sucres de surface des bactéries, font partie des vaccins les plus efficaces et les plus sûrs. Les vaccins glycoconjugués sont obtenus à partir de polysaccharides purifiés ou de mimes synthétiques de ces derniers couplés à une protéine dite protéine porteuse. A l’heure actuelle, ce procédé conduit à des mélanges complexes mal caractérisés.
Des approches de mutagenèse appliquées à la protéine porteuse nous ont permis de réaliser des couplages “site-spécifiques”, permettant l’obtention de conjugués homogènes, complètement définis. Ce résultat autorise une étude fine des relations structure/immunogénicité.
Les premiers résultats semblent indiquer que la position du sucre influe à la fois sur la réponse anti-sucre et sur la réponse dirigée contre les-épitopes B de la protéine porteuse.
Lorsque le sucre est placé sur ou à proximité d’un épitope B, il semble diminuer la réponse dirigée contre la protéine porteuse. Placer le sucre au niveau d’un épitope T auxiliaire permettrait d’obtenir une réponse anti-sucre plus importante.
Les approches de mutagenèse permettent d’envisager la préparation de vaccins tripartites constitués d’une protéine porteuse sur laquelle sont couplés non seulement le sucre de surface mais aussi des molécules adjuvantes. Dans cette optique, des analogues de l’α-galactosylcéramide, un puissant activateur des lymphocytes Natural Killer invariantes ont été synthétisés.
Titre: SulfAtlas: a classification database for Sulfatases reflecting their substrate specificity and its evolution.
L’instabilité génomique due à la dérégulation des voies de réparation de l’ADN peut être à l’initiation de cancer et entraîner par la suite une résistance à la chimiothérapie et à la radiothérapie. La compréhension de ces mécanismes biologiques est donc essentielle dans la lutte contre le cancer. RAD51 est la protéine centrale de la voie de réparation des cassures double-brin de l’ADN par recombinaison homologue. Cette réparation conduit à une réparation fidèle de l’ADN. L’activité recombinase de la protéine RAD51 est finement régulée par des modifications post- traductionnelles telles que la phosphorylation. Au cours de la dernière décennie, de plus en plus d’études, suggèrent l’existence d’une relation entre les récepteurs à activité tyrosine kinases, souvent suractivés et impliqués dans l’agressivité et la prolifération cancéreuse, et la réparation de l’ADN. Parmi ces récepteurs à activité tyrosine kinases, le duo c-Met/HGF-SF est souvent muté, sur exprimé ou activé constitutivement dans de nombreux cancers et son inhibition a été montrée comme induisant une diminution de la réparation par recombinaison homologue. Au travers de cette thèse, nous montrons pour la première fois que c-Met est capable de phosphoryler la protéine RAD51 sur quatre résidus tyrosine localisés principalement dans l’interface monomère- monomère du nucléofilament de la recombinase humaine. Nous montrons l’implication de ces phosphorylations sur l’activité de RAD51 dans les différentes étapes de la recombinaison homologue. L’ensemble des résultats obtenus suggère le rôle possible de ces modifications dans la régulation de RAD51 et souligne l’importance de c-Met dans la réponse aux lésions de l’ADN.
This study is of great importance for the advancement in the development of pneumococcal vaccines and the improvement of their efficacy. Indeed, researchers have succeeded in preparing and characterizing chitosan nanoparticles loaded with antigen, suitable for immunization.
The currently available pneumococcal vaccines are not effective in complete prevention of pneumococcal infections. To develop a better vaccine, first, “we were able to produce a glycoconjugate antigen (molecule resulting from the binding of carbohydrates to proteins or carbohydrates to lipids) based on a common pneumococcal vaccine. On the basis of the positive immune responses generated by the glycoconjugate antigen in mice, we further envisaged that their combination with the nanotechnologies would generate a vaccine with enhanced potential,” explains Maruthi Prasanna.
Following this approach, chitosan nanoparticles were used to encapsulate the glycoconjugate. Finally, the developed nanovaccines generated an enhanced immune response against both the protein and polysaccharide components when compared to the naked glycoconjugate.
La modélisation des structures protéiques en l’absence d’homologie est appelée modélisation sans matrice, modélisation libre ou modélisation ab initio. À ce jour, ce type de modélisation reste le plus grand défi du domaine.
Durant cette thèse, nous avons apporté une contribution méthodologique pour relever ce défi en explorant l’utilisation d’un alphabet structurel connu sous le nom de blocs protéiques (BP). Il est maintenant possible de prédire le squelette carbonné d’une protéine sous forme de séquence de BPs de n’importe quelle protéine en utilisant des outils comme PB-kPRED. Notre travail a consisté à trouver une méthode pour passer de la séquence de BPs prédite à la structure 3D.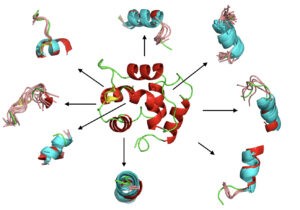
Nous avons donc développé une stratégie qui part d’une requête sous forme de séquences de BP prédites et recherche dans une base de données de structures non homologues des fragments de BPs similaires.
Notre stratégie tente ensuite d’assembler des fragments sélectionnés pour générer des modèles 3D. Pour cela, nous avons tenté d’exploiter les propriétés inhérentes de Modeller en la contraignant à utiliser des fragments de BPs et à utiliser des prédictions de structures secondaires et de contact comme contraintes externes supplémentaires.
Nous avons validé les résultats de cette stratégie sur un sous-ensemble de petites protéines cibles issues de CASP13 sur la base des scores couramment utilisés (GDT_TS et TM-score) pour les protocoles de modélisation gratuits. Nous avons pu montrer que des modèles de bonne qualité peuvent être générés pour de petites protéines en utilisant une combinaison de fragments à base de PB et de Modeller. Ce travail ouvre de nouvelles perspectives d’application des blocs protéiques pour solutionner l’épineux problème du repliement des protéines.
Au cours de cette thèse, nous avons mis en place une technologie d’incorporation d’acides aminés non naturels de manière site-spécifique au sein du laboratoire et l’avons appliqué à deux problématiques. Les vaccins glycoconjugués actuels ne couvrent que 13 des 97 sérotypes de Streptococcus pneumoniae. L’utilisation d’une protéine porteuse immunogène permet d’élargir la couverture sérotypique en couplant l’utilisation d’antigènes polysaccharidiques avec des antigènes protéiques. Cependant, les méthodes de conjugaison actuelles ne permettent pas l’étude de l’association optimale entre ces deux antigènes. Dans une première partie, nous avons généré différents vaccins glycoconjugués homogènes en incorporant la Propargyl-Lysine dans la protéine porteuse du pneumoncoque (mPsaA) afin de maîtriser la position ainsi que le nombre des haptens. Les lectines reconnaissent leur ligand saccharidique avec une très bonne spécificité mais avec une affinité généralement faible. Dans une seconde partie de la thèse, nous nous sommes intéressés à l’incorporation d’acides aminés non naturels permettant de former des liaisons covalentes avec les sucres et ainsi augmenter les forces d’interaction entre des lectines et leurs ligands. Nous avons tenté d’incorporer un premier acide aminé non naturel original sans succès. Mais deux autres ont pu être incorporés, dont l’un d’eux au sein de 2 cibles protéiques, en quantité suffisante pour réaliser des tests d’affinité. Cette thèse a donc permis la mise en place d’un nouvel axe technologique au sein du laboratoire et d’en explorer des applications potentielles.
Titre: Structure du filament RecA en solution analysee par des modelisations moleculaires et des mesures de SAXS.
Titre: Everything you always wanted to know about sex.. in diatoms.
Un ensemble de méthodes permettant d’étudier in silico des interactions protéine-glucide, primordiales dans de nombreux processus biologiques, sont proposées dans cette étude. En s’appuyant sur des informations fournies par la bio-cristallographie, il est devenu possible d’étudier à grande échelle les modalités d’interaction existantes entre ces deux entités moléculaires. Une étude statistique complète fut donc réalisée afin de déterminer aussi bien les tendances générales que les cas extrêmes observés dans les complexes protéine glucide. Les caractéristiques des interactions protéine glucide sont ainsi décrites, en particulier les liaisons hydrogène (LH) et le rôle des molécules d’eau. Un programme d’identification des LH et de reconstitution de leur(s) réseau(x) hypothétique(s) a été développé. Il comprend entre autres l’ajout putatif des hydrogènes, dans le cas où ils sont absents de la structure, notamment sur les groupes hydroxyles et les molécules d’eau. Une stratégie originale pour positionner de manière putative des molécules d’eau sur les sites de reconnaissance protéiques est également décrite. Cette stratégie a pour prétention de permettre le développement d’un protocole d’amarrage moléculaire protéine-glucide, les glucides et les molécules d’eau partageant sensiblement le même réseau de LH. Suite aux nombreuses anomalies décelées dans la PDB au niveau des glucides, une méthode d’identification et de vérification des structures 3D des glucides est également décrite. Elle a permis de limiter les bruits statistiques de cette étude. Environ 280 monosaccharides sous forme furanique et pyranique ont ainsi été référencés. La flexibilité intrinsèque des glucides a également amené à une étude approfondie de la conformation des glucides observée dans les structures cristallographiques.
Titre: Organization of bacterial cells: from chromosome dynamics via development of cell pole.
Even though their small size and lack of intercellular compartments, bacterial cells are also well organized. As bacteria grow by binary fission, cell division happens at the mid-cell which results in creating a new cell pole. Vibrio cholerae, curved Gram-negative rod that causes cholera, has several key cellular machines found at the old cell pole such as monotrichous flagellum, chemotactic array and the Par apparatus for segregating the larger chromosome (chrI). We previously identified the polar protein which tethers ParA1 chromosome partitioning protein to the cell poles. This protein was dubbed as HubP (hub of the pole), since it also anchors other proteins including ParC which recruits chemotaxis machinery and FlhG which involved in expression of flagella genes. Recent studies unveiled polar scaffolding proteins and their partners in many bacterial species.
Although these pole-organizing factors are diverse, bacteria appear to use overarching principles to arrange multiple macromolecular machineries to the specific site of the cell.
In this seminar, I will present our ongoing works to gain comprehensive view of organization at V. cholerae cell pole via HubP. First, we used comparative proteomics approach for further identification of polar proteins. We have identified 4 novel interaction partners of HubP: they include a penicillin binding protein and two proteins with unknown function. Lines of evidence indicated that the two new proteins are involved in cell motility. Second, we carry out super-resolution microscopy to define the precise spatial relationship among HubP, its interaction partners, and the cell pole. I will show our development of biological and informatics tools to integrate super-resolution microscopy data into multicellular analysis and succeeding investigation of precise localization.
Comprendre comment les mutations impactent l’activité d’une protéine reste un défi dans le domaine des sciences protéiques. Les méthodes biochimiques traditionnellement utilisées pour résoudre ce type de questionnement sont très puissantes mais sont laborieuses à mettre en œuvre. Des approches bioinformatiques ont été développées à cet égard pour surmonter ces contraintes. Dans cette thèse, nous explorons l’utilisation d’approches bioinformatiques pour comprendre le lien entre mutations et changements d’activité. Notre modèle d’étude est une enzyme bactérienne, la sucrose phosphorylase de Bifidobacterium adolescentis (BaSP). Cette glycosyl-hydrolase de la famille 13 (GH13) suscite l’intérêt de l’industrie en raison de sa capacité à synthétiser des disaccharides et des glycoconjugués originaux. Son activité consiste à transférer un glucose d’un donneur, le saccharose, à un accepteur qui peut être un monosaccharide ou un aglycone hydroxylé. La réaction enzymatique se déroule selon un mécanisme dit « double déplacement avec rétention de configuration », ce qui nécessite la formation d’un intermédiaire covalent dit glucosyl-enzyme. Cependant, la possibilité de contrôler la régiosélectivité de ce transfert pour qu’il soit applicable au niveau industriel est un enjeumajeur. Cette thèse vise d’une part, à fournir une explication rationnelle quant aux modifications de la régiosélectivité de BaSP apportées par des mutations et d’autre part à proposer un canevas pour le contrôle de la régiosélectivité de couplage en vue de la synthèse de disaccharides pré-biotiques rares comme le kojibiose et le nigerose. Dans notre approche, nous avons émis l’hypothèse que les orientations préférées de l’accepteur dans le site catalytique après formation du glycosyl-enzyme déterminent la régiosélectivité de l’enzyme. Nous avons utilisé des approches computationnelles pour étudier l’impact des mutations sur la liaison de l’accepteur à l’intermédiaire covalent, le glucosylenzyme. À cette fin, nous avons construit des modèles à l’échelle atomique du glucosyl-enzyme pour un ensemble de variants de la BaSP pour lesquels des données expérimentales étaient disponibles. Pour y parvenir, nous avons paramétré le glucosyl-aspartyle en tant que nouveau résidu et les avons intégré dans des outils de modélisation tels que Modeller et Gromacs. Nous avons évalué la pertinence de ces paramètres et les avons ensuite appliqués à la vérification de notre hypothèse de travail par le biais d’expériences d’ancrage moléculaire. La méthodologie utilisée dans ce travail ouvre la perspective de l’utilisation d’approches bioinformatiques pour l’ingénierie de la régiosélectivité de la sucrose phosphorylase et plus généralement des glycosylhydrolases possédant un mécanisme similaire. À cet égard, un pipeline de modélisation moléculaire et d’amarrage de molécules accepteurs sur des intermédiaires covalents des enzymes de cette famille (ENZO pour Optimisation d’ENZyme) a été développé au cours de cette thèse. Son application à l’ingénierie d’autres variants de BaSP est en cours.
Les agars sont des polysaccharides majeurs issus de la paroi des algues rouges. Ces polysaccharides sont composés par des résidus D- et L-galactose alternativement liés par des liaisons glycosidiques -1,4 et -1,3. Ces galactanes présentent de nombreuses modifications: sulfatations, méthylations, pyruvilation, branchement, etc.. Toutes ces modifications rendent complexe la dégradation de ces polysaccharides pour des bactéries marines hétérotrophes. Zobellia galactanivorans est une flavobactérie marine capable de dégrader de nombreux polysaccharides d’algues dont les agars. L’objectif de cette thèse était d’identifier et de caractériser les outils enzymatiques dont dispose cette bactérie pour dégrader les agars complexes. Les deux sujets d’étude de cette thèse sont une -agarase divergente (ZgAgaC) et des sulfatases actives sur des agars. Pour pouvoir réaliser cette étude portant sur des enzymes spécifiques des agars, nous avons mis au point des cribles d’activités sur une collection d’agars naturels qui a été préparée au cours de cette thèse. Ces cribles ont permis de mettre en évidence le comportement divergent de ZgAgaC sur des agars complexes, par rapport aux autres -agarases et -porphyranases de la famille 16 des glycosides hydrolases (GH16). Ils ont aussi permis d’identifier les agars complexes sur lesquels deux sulfatases différentes sont actives. En conclusion, ce travail de thèse a permis de mettre au point différents outils permettant de mettre en évidence de nouvelles activités enzymatiques et aussi de mieux comprendre le catabolisme des agars chez Z. galactanivorans.
—
Agars are red algal polysaccharides. These are composed of D-galactose with L-galactose alterned by glycosidic bond -1,4 and -1,3. These galactans harbor several modifications : sulfatations, methylations, pyruvylations. All these modifications hinder the agar degradation by marine bacteria. Zobellia galactanivorans is a marine flavobacterium able to degrade marine polysaccharides, including agars. The aims of this thesis are to identify and characterize the enzymatic tools of Z. galactanivorans to degrade the complex agar. The two subjects are a divergent -agarase (ZgAgaC) and sulfatases that act on agars. To perform this study on the agar specific enzymes, we have developed activity screens on a complex agar collection of substrates produced during the thesis. These screens have been used to show the divergent behaviour of ZgAgaC on complex agar compared to the other -agarases and -porphyranases of the family 16 (GH16). These screens were further used to identify the substrate of two sulfatases active on agar. To conclude, this work has allowed to develop different tools to identify new enzymatic activities and to have a better view of the agar catabolism of Z. galactanivorans.
Titre: Les mécanismes moléculaires de la perception des odeurs.
This thesis project covers the design, development and in vivo evaluation of a novel self-assembled injectable biodegradable drug delivery system (DDS), composed of in situ hydrogel combined with nanocapsules for lipophilic anti-inflammatory drugs, aiming prolonged intra-articular (IA) residence time and controlled drug release. The moderate initial viscosity allows good syringeability, while rheological properties of the assembled DDS enable resistance to high deformations, displaying the hydrogel suitable for IA application. In this regard, to validate the new delivery system, dexamethasone was used as a model drug. Besides, a novel and potential lead compound for immunotherapeutic anti-rheumatic drug candidate – galectin-3 antagonist was synthesized, characterized and evaluated in this study. The preliminary in vivo results demonstrated remarkable suppression of acute joint inflammation by galectin-3 inhibitor encapsulated within hydrogel and administrated IA at microgram scale doses compared to non-treated control. Overall, the work presented here portrays the DDS with encapsulated synthetic galectin-3 inhibitor as a capable in situ nanotechnology-based platform for arthropathies treatment, intended to contribute to efficient joints therapies.
Titre: Role of PML nuclear bodies in protein degradation and antiviral defense.
Titre: RecA nécessite deux molecules de Mg2+ pour son activité optimale.
Mg ion stimulates the DNA strand exchange reaction catalyzed by RecA, a key step in homologous recombination. To elucidate the molecular mechanisms underlying the role of Mg 2+ and the strand exchange reaction itself, we investigated the interaction of RecA with Mg 2+ and sought to determine which step of the reaction is affected. Thermal stability, intrinsic fluorescence, and native mass spectrometric analyses of RecA revealed that RecA binds at least two Mg 2+ ions with K D ≈ 2 mM and 5 mM.
Deletion of the C-terminal acidic tail of RecA made its thermal stability and fluorescence characteristics insensitive to Mg 2+ and similar to those of full-length RecA in the presence of saturating Mg 2+ . These observations, together with the results of a molecular dynamics simulation, support the idea that the acidic tail hampers the strand exchange reaction by interacting with other parts of RecA, and that binding of Mg 2+ to the tail prevents these interactions and releases RecA from inhibition. We observed that binding of the first Mg 2+ stimulated joint molecule formation, whereas binding of the second stimulated progression of the reaction. Thus, RecA is actively involved in the strand exchange step as well as bringing the two DNAs close to each other.
Titre: Genome and epigenome mediated regulation of microalgae success in contemporary oceans : insights from model species.
Les cellules humaines sont soumises à des stress induisant des cassures double-brin de l’ADN principalement réparées par la voie NHEJ, où la kinase DNA-PKcs joue un rôle central. L’activité de DNA-PKcs, régulée par de nombreuses phosphorylations, est cruciale pour le maintien de l’intégrité génomique. Plus récemment, il a été montré que cette protéine était également modifiée par l’O-GlcNAcylation dans la lignée COS7. Sachant l’équilibre existant entre phosphorylation et O-GlcNAcylation, nous avons étudié le rôle de cette nouvelle MPT dans la régulation de l’activité de DNA- PKcs. Nous avons montré que DNA-PKcs est O-GlcNAcylée dans les cellules HeLa. Puis nous avons montré que la modulation de l’O-GlcNAcylation de DNA-PKcs impacte son autophosphorylation en Ser2056, suggérant l’existence d’une balance O-GlcNAcylation /phosphorylation, ainsi que la capacité des cellules à réparer les DSBs par la voie NHEJ. De plus, nos résultats nous laissent envisager que cette modification puisse jouer un rôle dans la stabilité de la protéine. DNA-PKcs est une cible potentielle dans les stratégies de lutte contre le cancer. Nous avons étudié l’impact d’un composé sur DNA-PKcs. Cette molécule provoque une réduction de la quantité et de l’activité de DNA-PKcs, impliquant son ubiquitinylation et sa dégradation par le protéasome et menant à une sensibilisation des cellules à un traitement génotoxique. Dans ce contexte, nous avons développé une puce à anticorps pour évaluer le profil phosphoprotéique des voies de réparation de l’ADN et ainsi évaluer l’effet d’inhibiteurs de DNA-PKcs. L’ensemble de ces résultats contribuent à une meilleure compréhension de la régulation de DNA-PKcs.
Structural motifs found in protein structures. They help in approximating protein structure as a 1D string with minimal loss of structural information. Here I have employed a widely used structural alphabet called Protein Blocks (PB) for various applications like predicting, comparing and analyzing protein structures. PBs were used to study the structural variations in proteins with identical primary structure and also during the course of molecular dynamics (MD) simulations. The results from these analyses were summarized in the form of substitution matrices and showed striking similarities to previously established matrices for homologous proteins and NMR ensembles. I improved kPRED, a knowledge-based prediction of protein backbone in terms of PBs, by taking into consideration the neighboring local structures. The new version of the algorithm also privileges structural information from homologous proteins when available reaching an average accuracy of 66.3% on a benchmark dataset. The scope of the PENTAdb database has been expanded to cover the entire protein structure space in an automated manner. I show that the effect of this 950% increase in the contents of PENTAdb improves our understanding of the sequence-structure relationship at the pentapeptide level. I also used PB-ALIGN, a fast and efficient protein structure comparison tool, to compare all protein structures in PDB in an all-vs-all manner and to investigate PB-based structural similarities. This generated a huge collection of alignment data and I discuss its use for functional annotation and identification of possible evolutionary relationships.
La protéine RAD51 est l’un des acteurs majeurs des mécanismes de la résistance et de la reconstruction des cellules cancéreuses. La modulation de son activité ouvre une nouvelle voie dans la lutte contre le cancer. De nouveaux inhibiteurs potentiels de RAD51 ont été recensés, notamment, les dérives du stilbène disulfonique (SD). Dans cette thèse, nous étudions ces inhibiteurs dans différents milieux (solvant et protéine). La première partie de cette thèse est dédiée à l’étude des propriétés physico-chimiques en solution des dérivés SD. À l’aide de spectres optiques expérimentaux et de simulations par calculs quantiques (DFT et TD-DFT), nous avons montré que les SD se présentent sous leur forme dianionique en condition physiologique et la prise en compte du couplage vibronique est cruciale pour simuler des bandes des spectres d’absorption. La seconde partie se focalise sur la modélisation du complexe SD2- en interaction avec la protéine de transport albumine sérique humaine (ASH), qui joue un rôle prépondérant dans le transport de molécule au sein du corps humain. Elle apparaît être un excellent candidat pour acheminer des inhibiteurs SD2- vers les cellules cancéreuses. Sans donnée cristallographique du complexe ASH-SD2-, la modélisation moléculaire est le seul outil prédictif utilisable pour obtenir des données structurales, et nous avons associé ici des méthodes de docking moléculaire et de dynamique moléculaire. Cette méthodologie nous a permis (i) d’identifier le(s) résidu(s) important(s), (ii) d’évaluer les énergies d’interaction par des calculs MM-GBSA et (iii) d’identifier les sites d’accueil les plus adéquats pour les composés de type SD2-.
Titre: Discovery, design and development of human amylase inhibitors : from nM to pM.
Carbohydrates play important roles in biological systems, not only in the form of energy storage materials such as starch, but also as “recognition elements” on cell surfaces. The degradation of such sugar structures is achieved using enzymes known as glycoside hydrolases (glycosidases). Specific enzyme inhibitors are not only useful tools for understanding enzyme mechanisms, but also can play important roles as therapeutics if inhibition suppresses unwanted reactions. In this talk I shall discuss our efforts to discover new inhibitors of human pancreatic alpha-amylase (HPA) that have potential in the control of blood sugar levels, thus in the treatment of diabetes and possibly obesity.
High-throughput screening of natural product extract libraries from terrestrial and marine sources, in conjunction with my colleague Ray Andersen, has yielded two new classes of potent (Ki = 8 nM and 10 pM) inhibitors of human α-amylase. The first compound, Montbretin A, was isolated from the beautiful Crocosmia plant shown below, and its inhibition characterized through degradation studies and through X-ray crystallographic analysis of its complex with HPA in conjunction with Gary Brayer [1]. Subsequent animal studies revealed, good control of blood glucose levels in diabetic rats when administered orally [2]. The second screen revealed helianthamide as a highly potent peptidic inhibitor of HPA, derived from a sea anemone [3].
Structural studies with these inhibitors reveal a new paradigm for glycosidase inhibition in which pairs of aromatic moieties joined via a short linker provide the key inhibitory motif. Synthesis of simple mimics of this core structure has yielded inhibitors with Ki values ranging down to 50 nM to date, while screening of very large libraries of cyclic peptides through ribosome display methodologies in conjunction with the Suga group has produced two sets of inhibitors with Ki values between 1 and 10 nM, with similar modes of inhibition [4].
1. Williams, L. et al (2015) Nature Chemical Biology 11, 691-696.
2. Yuen, V.G. et al 2016) Mol. Cell. Biochem. 411, 373-381.
3. Tysoe et al (2016) ACS Central Science 2, 154-161.
4. Jongkees et al (2017) Cell Chemical Biology 24, 381-390.
Catalyseurs de la dégradation de polysaccharides dans le cadre de diverses applications industrielles, de nombreuses glycoside hydrolases (GH) possèdent également une activité de transglycosylation qui peut être exploitée pour la synthèse d’oligosaccharides. Afin d’augmenter cette activité, minoritaire par rapport à l’hydrolyse, des expériences de mutagenèse rationnelle peuvent être employées. Toutefois, l’ensemble des bases moléculaires régissant l’équilibre entre ces deux activités reste en revanche difficile a élucider. L’étude de quatre GH (Ttβgly, AgaD, TcTS, TrSA) par simulation de dynamique moléculaire a permis la découverte de canaux d’eau internes à leurs structures et connectant le site actif au milieu. Cette observation suggère que les canaux d’eau internes aux GH pourraient être impliqués dans leur activité d’hydrolyse. Plusieurs paires de résidus bordant deux de ces canaux ont été mis en évidence chez Ttβgly et AgaD et semblent contrôler le passage de l’eau du canal vers le site actif. La mutagenèse de ces résidus a été entreprise afin de tenter d’augmenter l’activité de transglycosylation chez ces deux enzymes. Une réduction de l’hydrolyse d’un facteur 7 et 50 au profit de l’activité de transglycosylation a été caractérisée chez les deux meilleurs mutants de Ttβgly et AgaD, respectivement. L’analyse des simulations a révélé que ces résultats étaient corrélés à une augmentation de la dynamique des molécules d’eau internes aux deux canaux étudiés. Cette étude souligne ainsi l’importance fonctionnelle de l’eau interne aux hydrolases et suggère que l’ingénierie de sa dynamique peut constituer une approche originale pour convertir les GH en transglycosidases.
Omniprésentes dans le règne animal et végétal, les galectines occupent de nombreuses fonctions biologiques au sein des organismes vivants. Cette famille de lectines, capable de se lier aux B-galactosides grâce à un domaine de reconnaissance des sucres conservé, compte quinze membres chez les mammifères. Ces protéines ont pu être identifiées comme des acteurs importants de plusieurs processus biologiques tels que le cycle cellulaire, la réponse immunitaire ou encore la migration cellulaire. Plus récemment les galectines ont été identifiés comme des cibles thérapeutiques de choix car la dérégulation de l’expression de ces protéines a pu être corrélée à plus d’une centaine de pathologies (cancer, diabète, maladie inflammatoire,…). L’un des enjeux majeurs concernant la recherche sur ces protéines, est de parvenir à synthétiser des outils permettant une meilleure compréhension de leurs rôles au sein de l’organisme. Portant une attention plus particulière à la galectine-3, nous avons mis au point des voies de synthèses permettant l’obtention d’inhibiteurs affins et sélectifs de cette dernière, et ce dans le but d’être utilisés comme outils dans l’étude du rôle des galectines dans le phénomène de migration cellulaire des cellules épithéliales de la peau. Appliquant une réaction d’azido-phenylsélénation au lactal ou par une approche chimio-enzymatique, nous avons pu synthétiser deux familles de nouveaux composés dérivés de la lactosamine. L’affinité et la sélectivité de ces composés vis-à-vis des galectines ont été déterminées par des techniques de bio-puces, de polarisation de fluorescence ou encore de microcalorimétrie.
Les cellules humaines sont soumises à des stress induisant des cassures double-brin de l’ADN (CDB). Ces CDB sont réparées notamment par la recombinaison homologue, impliquant les protéines RAD51 et RAD52. Une stratégie thérapeutique émergente est de développer des molécules inhibant RAD51 ou RAD52 afin d’accentuer l’instabilité génétique et la mort de la cellule cancéreuse. En effet, dans certains cancers, l’activité de RAD51 est dérégulée promouvant la prolifération tumorale. Il existe plusieurs molécules inhibitrices de RAD51 et nous nous sommes intéressés au DIDS dont le mode d’action n’a pas encore été déterminé. Concernant RAD52, une létalité synthétique a été montrée lorsque celle-ci est inactivée dans des cellules déficientes en BRCA1, BRCA2 ou PALB2, trois gènes mutés dans de nombreux cancers. Récemment, trois types de molécules inhibitrices de RAD52 ont été mis en évidence. Nous avons tout d’abord étudié l’impact du DIDS ainsi que des molécules dérivées afin de comprendre le mécanisme mis en jeu. Nous avons montré que le DIDS, ainsi que ses dérivés inhibent la liaison de RAD51 à l’ADN. Ces molécules empêchent la formation du nucléofilament entrainant une diminution du nombre de foyers RAD51. Nous avons développé une méthode de criblage par fluorescence pour évaluer l’effet d’une banque de 696 molécules sur la capacité de RAD52 à hybrider deux ADNsb. Deux molécules capables d’inhiber la fonction d’hybridation de RAD52 ont été mises au jour. In vivo, elles entrainent une diminution de la survie de cellules déficientes en PALB2. La recherche et le développement de nouveaux inhibiteurs de RAD51 et RAD52 constituent des stratégies thérapeutiques d’avenir.
La tomographie par émission de positon (TEP) est une technique d’imagerie médicale permettant le diagnostic et le suivi de patient en oncologie. L’utilisation des anticorps et de leurs fragments pour le ciblage de biomarqueurs en TEP présente de nombreuses difficultés liées notamment à leur clairance lente (taille >100 kDa). Leur marquage, faisant intervenir principalement des réactions peu spécifiques, conduit à un mélange hétérogène de produits et parfois à l’inactivation des protéines.
Le développement d’un nouvel outil de suivi in vivo des patients à l’aide de petites protéines alternatives aux anticorps, les Nanofitines (NF), permet de s’affranchir des contraintes liées à la taille (NF ≈ 10 kDa). La mise en place d’une stratégie de marquage originale et site- spécifique d’une NF sans étape de couplage chimique a d’abord été envisagée dans cette étude.
L’approche est basée sur la capacité naturelle des phosphates à fixer des cations métalliques. L’insertion génétique d’une étiquette peptidique phosphorylable, in vivo ou in vitro, a permis la chélation d’un lanthanide en solution, le Tb(III), avec une affinité de l’ordre du μM. La seconde génération d’étiquettes peptidiques obtenues par mutagenèse a permis la chélation du Tb(III) avec une affinité d’environ 500 nM à pH7 et 50 nM à pH5,5, et une affinité pour le Ga(III) de l’ordre du μM à pH5,5. Parallèlement, la biodistribution et le ciblage spécifique in vivo d’une NF anti-EGFR radiomarquée à l’aide du 18F-FBEM ont été évalués dans un double modèle tumoral murin. Les images TEP obtenues avec un bon contraste ont permis de valider la preuve de concept quant à l’utilisation des NF en tant qu’outil en imagerie médicale.
La Recombinaison Homologue (RH) permet la réparation des dommages à l’ADN les plus délétères : les Cassures double brin. L’étape centrale de la RH est basée sur l’activité d’échange de brins de RAD51. Ainsi, l’activité de RAD51 est cruciale pour le maintien de l’intégrité génomique. Toutefois, cette protéine possède également un côté sombre. En effet, la surexpression de RAD51 permet aux cellules cancéreuses de résister aux traitements. Ce qui en fait une cible thérapeutique potentielle pour sensibiliser les cellules cancéreuses au traitement. Une meilleure compréhension du contrôle de l’activité de RAD51 aiderait sûrement à développer des stratégies thérapeutiques. L’activité de RAD51 est régulée par des phosphorylations et plusieurs kinases sont connues pour cibler RAD51. C’est le cas de la kinase c-Abl qui phosphoryle les tyrosines Y54 et Y315 en réponse aux dommages à l’ADN. Mais le rôle de ces phosphorylations est peu connu. C’est pourquoi nous nous sommes intéressés à l’effet de ces phosphorylations sur RAD51. Dans ce but, nous avons produit des mutants de RAD51 mimant la phosphorylation. Leur activité a été analysée et comparée in vitro. Nous avons démontré que le mutant équivalent à une double phosphorylation est incapable de réaliser l’échange de brins. Un défaut de polymérisation de RAD51 serait à l’origine de cette inhibition. Par la suite, la régulation a été étudiée dans le contexte cellulaire. Les résultats préliminaires montrent un effet de la double phosphorylation sur la localisation cellulaire de RAD51. L’inactivation de RAD51 par cette double phosphorylation pourrait participer à la régulation de la voie de la Recombinaison Homologue et serait une étape clef dans la compréhension de la réponse aux dommages à l’ADN
La reconnaissance moléculaire, ou formation spécifique de complexes entre molécules par l’intermédiaire d’interactions physico-chimiques, est un mécanisme d’action fondamental des protéines appliquées à un usage médical. Dans cette étude, nous explorons l’ingénierie rationnelle de cette fonction avec comme partenaire une nouvelle classe de protéines destinée à développer les médicaments de demain: les Nanofitines (NF). Dans l’objectif de guider les approches expérimentales (in vitro) par celles prédictives (in silico) dans une collaboration réciproque, ce manuscrit décrit successivement deux preuves de concept appliquées à des NF dirigées contre la protéine de fluorescence verte (GFP). La première approche, conçue pour l’humanisation de NF par transfert sur des protéines humaines, a consisté à isoler 11 résidus fonctionnels à greffer à partir de NF anti-GFP générées par ribosome display. Le squelette du feuillet beta exposant ces acides aminés clés a ensuite été recherché dans une banque structurale pour identifier trois protéines humaines hôtes arborant un feuillet similaire (RMS ≤ 0,63Å). Une fois greffées des résidus clés des NF, ces protéines ont démontré une insolubilité ou une absence de fixation détectable à la GFP, contrairement à un homologue structural des NF. La seconde approche a visé à réaliser le design de novo de NF anti-GFP par modélisation moléculaire. Cette démarche, proposée comme alternative rationnelle complémentaire du procédé de sélection in vitro, a abouti à la découverte d’une NF dont l’épitope ciblé s’avère compatible avec les prédictions réalisées. Cette stratégie nécessitera d’être complétée afin d’optimiser l’affinité des NF générées de novo.
L’hydrogène est un candidat viable pour la prochaine génération de carburant grâce à sa den- sité d’énergie élevée et à son caractère non polluant lors de la phase de production d’énergie. La production biologique à partir de microorganismes capable de synthétiser l’hydrogène, souffre actuellement de faibles rendements de production car les cellules doivent maintenir différentes activités cellulaires, autres que la production d’hydrogène, afin de survivre. Pour répondre à ce problème, des chercheurs ont exploré l’assemblage synthétique d’enzymes in vitro dans des systèmes acellulaires ayant des fonctions spécifiques. Un tel système synthétique acellulaire a été conçu en combinant 13 différentes enzymes pour la synthèse de l’hydrogène à partir de différents composés cellulosiques dont le cellobiose. Cet assemblage a un meilleur rendement que les systèmes basés sur l’exploitation de microorganismes. Nous avons utilisé des méthodes basées sur la résolution d’équations différentielles pour étudier comment les conditions initiales et les paramètres cinétiques des enzymes influençaient la productivité d’un tel système, et pour identifier, à travers des simulations, les conditions optimisant la production d’hydrogène à partir de cellobiose comme substrat. En outre, si les paramètres cinétiques des enzymes constitutives d’un tel système ne sont pas connus, nous avons montré comment, en utilisant des réseaux de neurones artificiels, il est possible d’identifier d’autres modèles qui permettent d’avoir une idée de la cinétique de la production d’hydrogène. Au cours de notre étude sur le système utilisant le cellobiose, d’autres assemblages acellulaires ont été conçus pour produire de l’hydrogène à partir de différentes matières premières. Intéressé dans la reconstruction de systèmes synthétiques, nous avons décidé de concevoir divers outils pour aider à l’automatisation de l’assemblage et de la modélisation de ces nouveaux réseaux synthétiques. Ce travail démontre comment la modélisation peut aider dans la conception et la caractérisation des systèmes acellulaires en biologie synthétique.



