Responsable : Yves-Henri SANEJOUAND
L’équipe “Bioinformatique Structurale” a deux objectifs principaux :
- Développer des méthodes permettant d’étudier la relation séquence-structure-dynamique-fonction des protéines, en privilégiant des approches originales (modes normaux, alphabet structural…). Plus particulièrement:
- la relation séquence-structure: des alphabets structuraux sont développés, notamment dans le but de prédire des structures locales et tertiaires (problème de la reconnaissance de repliements).
- la relation structure-flexibilité: l’analyse en modes normaux est utilisée pour produire des jeux de conformères de protéines capables de récapituler ceux observés lors de simulations de dynamique moléculaire, ainsi que pour évaluer l’importance du rôle de l’énergie libre vibrationnelle dans les phénomènes de reconnaissance moléculaire.
- la relation structure-fonction : cette relation est notamment étudiée dans le cadre de nos travaux consacrés aux phénomènes de reconnaissance protéine-molécule odorante.
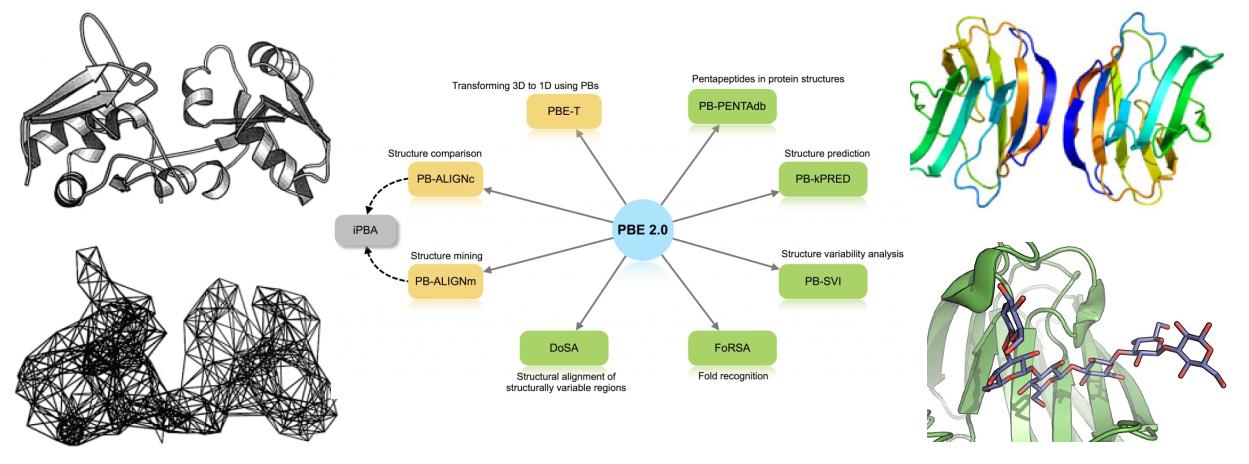
Les méthodes développées sont mises à disposition de la communauté sous forme de sites web : Protein Blocks Expert, Elnémo, etc.
- Contribuer aux interprétations de résultats expérimentaux obtenus par nos collaborateurs. C’est l’occasion pour nous de tester des méthodes standards dans des cas autres que les systèmes modèles les plus populaires de notre domaine, telles que les simulations de dynamique moléculaire classique, les méthodes d’arrimage protéine-ligand ou protéine-protéine. Nous travaillons notamment sur les complexes protéine-(oligo)saccharide, en collaboration avec l’équipe de Glycobiologie. Le problème de la reconnaissance protéine-protéine est quand à lui abordé dans le cadre d’une importante collaboration régionale (le projet PIRAMID).
Les analyses effectuées sont mises à disposition de la communauté sous forme de sites web : dockNmine, Proteocarb, etc.
Mots-clés : alphabet structural, bioinformatique, bioinformatique structurale, docking, interactions protéines/sucres, machine learning, modélisation moléculaire, modes normaux, reconnaissance moléculaire
Membres
Anciens membres de l'équipe
- Eléna ALVAREZ SANCHEZ, Stagiaire
- Nicolas ANTUNES, Stagiaire
- Philippe ARNAUD, Ingénieur de recherche
- Hamady BA, Stagiaire
- Thierno Sidy BAH, Stagiaire
- Maximilien BERNE, Stagiaire
- Jérôme BOBE, Stagiaire
- Cynthia BORES, Stagiaire
- Charlotte BRUNEL-CADIC, Stagiaire
- Lucie CARTAIRADE, Stagiaire
- Pierrick CHABOT, Stagiaire
- Louis CLOSSON, Stagiaire
- Anna CORNUAULT, Stagiaire
- Gwendoline COUTURIER, Stagiaire
- Pauline DACLIN, Stagiaire
- Benoît DAVID, Doctorant
- Lucas DAVID, Stagiaire
- Surbhi DHINGRA, Doctorante
- Annelise DMYTRYK, Stagiaire
- Marwane DRIBEK, Stagiaire
- Florian ECHELARD, Stagiaire
- Matthieu EVAIN, Stagiaire
- Nicolas FONTAINE, Doctorant
- Damien GARCIA, Stagiaire
- Ennys GHEYOUCHE, Doctorant
- Ronan GOUDE LE HENAFF, Stagiaire
- Ilyas GRANDGUILLAUME, Stagiaire
- Chloé GRIVAUD, Stagiaire
- Ipek GUVENTÜRK, Stagiaire
- Ollo Franck HIEN, Stagiaire
- Anne-Emeline HUARD, Stagiaire
- Simon HUET, Doctorant
- César HUNAULT, Stagiaire
- Stéphane JEDELE, Stagiaire
- Antoine LABEEUW, Stagiaire
- Romain LAUNAY, Stagiaire
- Aymeric LE NIR, Stagiaire
- EMIE LEFEVRE, Stagiaire
- Thibaud LEPOIVRE, Stagiaire
- Jean LETHIEC, Stagiaire
- Julien LOUET, Stagiaire
- Swapnil MAHAJAN, Post-doctorant
- Elvis MARTIS, Post-doctorant
- Pierre MICHON-NATIEZ, Stagiaire
- Augustin MOREAU, Stagiaire
- Chaïmae NACHAOUI, Stagiaire
- Gleb NOVIKOV, Post-doctorant
- Solène OKOM NDONG TATY, Stagiaire
- Sara PENARANDA, Ingénieure de recherche
- Steven PICHON, Stagiaire
- Justine PLOTEAU, Stagiaire
- Damien RAT, Stagiaire
- Dylan REMY, Stagiaire
- Dylan REMY, Stagiaire
- Tristan RIALLAND, Stagiaire
- Maëva RODRIGUEZ, Stagiaire
- Caroline ROZE, Stagiaire
- Timothée SALZAT HERVOUETTE, Stagiaire
- Dylan SERILLON, Stagiaire
- Rahamia SOILIHI MLANAOINDROU, Stagiaire
- Vinh TRAN, Professeur émérite
- Mahesh VELUSAMY, Doctorant
- Iyanar VETRIVEL, Doctorant
- Mallaury VIE, Stagiaire
Projets
Anciens projets
Publications
2 publications
Álvarez-Sánchez, Elena; Huet, Simon; Téletchéa, Stéphane
Molecular determinants of TNFR1:TNFα binding and dynamics in a physiological membrane environment Article de journal
Dans: Current Research in Structural Biology, vol. 26, p. 100177, 2026.
@article{TeletcheaTNF2025,
title = {Molecular determinants of TNFR1:TNFα binding and dynamics in a physiological membrane environment},
author = {Elena Álvarez-Sánchez and Simon Huet and Stéphane Téletchéa},
editor = {Elsevier},
doi = {10.1016/j.crstbi.2025.100177},
year = {2026},
date = {2026-06-01},
urldate = {2025-12-18},
journal = {Current Research in Structural Biology},
volume = {26},
pages = {100177},
abstract = {Tumor Necrosis Factor alpha (TNFα) is a pro-inflammatory cytokine critical for regulating cell survival and death. Under pathological conditions, excessive TNFα activity can lead to chronic inflammation, contributing to diseases such as inflammatory bowel disease and other autoimmune disorders. While structural studies have elucidated the atomistic details of TNFα binding to its receptor, TNF Receptor 1 (TNFR1), the influence of the membrane environment on this interaction remains poorly characterized experimentally. In this study, we employed advanced all-atom Gaussian accelerated molecular dynamics simulations to investigate how lipid-mediated interactions modulate the TNFα–TNFR1 complex. We identified key residues on both the cytokine and its receptor that govern trimer assembly, receptor binding, and potential pathological alterations. Our analysis confirmed previously identified functional sites and revealed new residues likely to contribute to the structural stability and dynamics of the complex. These findings provide a more comprehensive understanding of the molecular determinants of TNF signaling and offer a foundation for future experimental investigations into the receptor-ligand interface and membrane-mediated regulation.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Álvarez-Sánchez, Elena; Offmann, Bernard; Huet, Simon; Téletchéa, Stéphane
Energetics Decomposition of Sac7d:DNA Decrypts Amino Acids Role Without DNA Sequence Selectivity Article de journal
Dans: Journal Of Molecular Recognition, vol. 39, iss. 1, p. e70021, 2026.
@article{TeletcheaSac7d2025,
title = {Energetics Decomposition of Sac7d:DNA Decrypts Amino Acids Role Without DNA Sequence Selectivity},
author = {Elena Álvarez-Sánchez and Bernard Offmann and Simon Huet and Stéphane Téletchéa},
editor = {Wiley},
doi = {10.1002/jmr.70021},
year = {2026},
date = {2026-01-05},
urldate = {2025-12-15},
journal = {Journal Of Molecular Recognition},
volume = {39},
issue = {1},
pages = {e70021},
abstract = {Sac7d is a 7 kDa protein belonging to the class of the small chromosomal proteins from archeon Sulfolobus acidocaldarius. Sac7d was discovered in 1974 in Yellowstone National Parks geysers, and studied extensively since then for its remarkable stability at large pH and temperature ranges. Sac7d binds to the DNA minor groove, thereby protecting the host genome from extreme conditions by increasing the DNA melting temperature. In this study, we analyzed the Sac7d-DNA complex using 1 μs molecular dynamics simulations. The interaction energy of the interface was decomposed using Molecular Mechanics with Generalized Born Surface Area (MM/GBSA) to determine the residues that contributed most significantly to DNA binding. Out of 12 amino acids considered essential for DNA binding, three were newly identified in this study and had not been previously reported. One of these new amino acids, R63, may be involved in a dynamic protein-DNA interaction. The simulations performed also revealed a sliding motion of Sac7d over double-stranded DNA, suggesting a minimal sequence dependence interaction. Our analysis thus provides novel insights into how the Sac7d chaperones allow to protect DNA from degradation in extreme conditions. },
keywords = {},
pubstate = {published},
tppubtype = {article}
}
8 publications
Maillet, Laurent; Fétiveau, Aurélie; Lalier, Lisenn; Martin, Nena; Barillé-Nion, Sophie; Guette, Catherine; Gautier, Fabien; Téletchéa, Stéphane; Juin., Philippe Paul
Allosteric regulation of BH3-in-groove interactions by tail anchors of BCL-xL complexes limits BH3 mimetic antagonism Article de journal
Dans: Nature Commununications, vol. 16, iss. 1, p. 10621, 2025.
@article{TeletcheaNatCom2025,
title = {Allosteric regulation of BH3-in-groove interactions by tail anchors of BCL-xL complexes limits BH3 mimetic antagonism },
author = {Laurent Maillet and Aurélie Fétiveau and Lisenn Lalier and Nena Martin and Sophie Barillé-Nion and Catherine Guette and Fabien Gautier and Stéphane Téletchéa and Philippe Paul Juin. },
editor = {Nature},
doi = {10.1038/s41467-025-65509-1},
year = {2025},
date = {2025-11-22},
urldate = {2025-11-22},
journal = {Nature Commununications},
volume = {16},
issue = {1},
pages = {10621},
abstract = {BCL-xL promotes cell survival by binding BH3-only initiators through its hydrophobic groove. Combining resonance energy transfer assays and molecular dynamics simulations, we unravel that membrane anchoring of BCL-xL via its tail anchor selectively advantages binding to membrane-anchored PUMA initiator over BH3 mimetic ligands of the groove. This is due to the combined allosteric effect on BH3-in-groove binding of BCL-xL and PUMA tail anchors. Moreover, doubly anchored PUMA / BCL-xL complexes recruit endogenous BAX, which favors their antagonism by BH3 mimetics. BAX’s tail anchor alone is sufficient to enhance BH3 mimetics-induced death in cells expressing PUMA / BCL-xL. Our work supports a model in which the survival function of BCL-xL is regulated by a complex interplay between its tail anchor and those of its interacting partners. This enables both resistance to pharmacological inhibitors and modulation by BAX, which functions as a crucial feedback disruptor of the BCL-xL network.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Dhingra, Surbhi; Téletchéa, Stéphane; Sowdhamini, Ramanathan; Sanejouand, Yves-Henri; Brevern, Alexandre G.; Cadet, Frédéric; Offmann, Bernard
Using protein blocks to build custom fragment libraries from protein structures Article de journal À paraître
Dans: Biochimie, À paraître, ISSN: 0300-9084.
@article{DHINGRA2025,
title = {Using protein blocks to build custom fragment libraries from protein structures},
author = {Surbhi Dhingra and Stéphane Téletchéa and Ramanathan Sowdhamini and Yves-Henri Sanejouand and Alexandre G. Brevern and Frédéric Cadet and Bernard Offmann},
url = {https://www.sciencedirect.com/science/article/pii/S0300908425001907},
doi = {https://doi.org/10.1016/j.biochi.2025.08.011},
issn = {0300-9084},
year = {2025},
date = {2025-08-13},
urldate = {2025-01-01},
journal = {Biochimie},
abstract = {The remarkable structural diversity of modern proteins reflects millions of years of evolution, during which sequence space has expanded while many structural features remain conserved. This conservation is evident not only among homologous proteins but also in the recurrence of supersecondary motifs across unrelated proteins, underscoring the abundance and robustness of these structural units. Here, we present a novel pipeline for generating customized protein fragment libraries using protein blocks (PBs)—a structural alphabet that encodes local backbone conformations. Our method efficiently extracts structurally similar fragments from a curated, non-redundant protein structure database by converting three-dimensional structures into one-dimensional PB sequences. By integrating predicted PB sequences with the PB-ALIGN and PB-kPRED tools, our approach identifies relevant fragments independently of sequence homology. Fragment quality is further assessed using a new scoring function that combines secondary structure similarity and PB alignment metrics. The resulting libraries contain fragments of at least seven PBs (11 amino acid residues), covering over 70% of the local backbone structure. Our results demonstrate that PBs enable efficient mining of high-quality structural fragments from diverse protein spaces, including proteins with disordered regions. The pipeline is accessible as an online tool (PB-Frag, http://pbpred-us2b.univ-nantes.fr/pbfrag).},
keywords = {},
pubstate = {forthcoming},
tppubtype = {article}
}
Offmann, Bernard; Brevern, Alexandre G.
A 25-year journey with protein blocks: Unveiling the versatility of a structural alphabet Article de journal
Dans: Biochimie, 2025, ISSN: 0300-9084.
@article{OFFMANN2025,
title = {A 25-year journey with protein blocks: Unveiling the versatility of a structural alphabet},
author = {Bernard Offmann and Alexandre G. Brevern},
url = {https://www.sciencedirect.com/science/article/pii/S0300908425001750},
doi = {https://doi.org/10.1016/j.biochi.2025.08.007},
issn = {0300-9084},
year = {2025},
date = {2025-08-09},
urldate = {2025-01-01},
journal = {Biochimie},
abstract = {Protein Blocks (PBs) represent a widely used structural alphabet that enables the approximation and analysis of local protein conformations through 16 prototype fragments defined by dihedral angles. Initially developed to overcome the limitations of classical secondary structure definitions, PBs provide a powerful tool for understanding protein structure, dynamics, and function. Their applications span structural annotation, protein fold superimposition and recognition, sequence-based prediction and molecular dynamics analysis. Notably, PBs facilitate the distinction between rigid, flexible, and disordered regions via an entropy-based index (Neq), offering insights into protein flexibility and intrinsic disorder. Their integration with deep learning has dramatically improved predictive performance, and their utility has been demonstrated in diverse contexts such as integrin polymorphisms, VHH variability and AlphaFold structure analysis. As a robust and adaptable framework, PBs remain central in modern structural bioinformatics.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Fredslund, Folmer; Goux, Marine; Offmann, Bernard; Demonceaux, Marie; André-Miral, Corinne; Welner, Ditte; Teze, David
Crystal structure of the sucrose phosphorylase from Alteromonas mediterranea shows a loop transition in the active site Article de journal
Dans: Acta Crystallographica Section F, vol. 81, no. 7, p. 306–310, 2025.
@article{Fredslund:us5158,
title = {Crystal structure of the sucrose phosphorylase from Alteromonas mediterranea shows a loop transition in the active site},
author = {Folmer Fredslund and Marine Goux and Bernard Offmann and Marie Demonceaux and Corinne André-Miral and Ditte Welner and David Teze},
url = {https://doi.org/10.1107/S2053230X25004327},
doi = {10.1107/S2053230X25004327},
year = {2025},
date = {2025-07-01},
urldate = {2025-07-01},
journal = {Acta Crystallographica Section F},
volume = {81},
number = {7},
pages = {306–310},
abstract = {Sucrose phosphorylases are essential enzymes regulating sucrose metabolism, and it has been shown that a loop rearrangement is essential to their catalytic cycle. Crystal structures of only six sucrose phosphorylase enzymes are available. Here, we present the crystal structure of a sucrose phosphorylase from a proteobacterium, ıt Alteromonas mediterranea, at 2.15Å resolution. The available sucrose phosphorylase structures have shown that an important conformational change occurs during the catalytic cycle or upon mutagenesis. Interestingly, our data present clear indications of the two major conformations in the same crystal.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Martis, Elvis A F; Téletchéa, Stéphane
Ten quick tips to perform meaningful and reproducible molecular docking calculations Article de journal
Dans: PLoS Comput Biol, vol. 21, no. 5, p. e1013030, 2025, ISSN: 1553-7358.
@article{pmid40344147,
title = {Ten quick tips to perform meaningful and reproducible molecular docking calculations},
author = {Elvis A F Martis and Stéphane Téletchéa},
doi = {10.1371/journal.pcbi.1013030},
issn = {1553-7358},
year = {2025},
date = {2025-05-01},
urldate = {2025-05-01},
journal = {PLoS Comput Biol},
volume = {21},
number = {5},
pages = {e1013030},
abstract = {Molecular docking is a useful method for predicting the binding affinity and conformation of small chemical entities to support lead optimisation. It is also used to virtually screen a large chemical database to find new chemical entities. There are several docking programs available with different algorithms and varying preparation steps. We identify ten quick tips that apply to molecular docking irrespective of the program one might choose. Our objective is to provide the beginners with important things to keep in mind while using molecular docking for their research. We aim to ensure that experts and beginners can perform molecular docking to yield biologically relevant and reproducible results.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Téletchéa, Stéphane; Lombard, Bérangère; Hendrickx, Johann; Loew, Damarys; Tirichine, Leïla
Glutamate Methylation, a Novel Histone Mark in Diatoms: Mass Spectrometry Identification and Structural Characterization Article de journal
Dans: Plant Direct, vol. 9, no. 5, p. e70051, 2025, ISSN: 2475-4455.
@article{pmid40365482b,
title = {Glutamate Methylation, a Novel Histone Mark in Diatoms: Mass Spectrometry Identification and Structural Characterization},
author = {Stéphane Téletchéa and Bérangère Lombard and Johann Hendrickx and Damarys Loew and Leïla Tirichine},
doi = {10.1002/pld3.70051},
issn = {2475-4455},
year = {2025},
date = {2025-05-01},
urldate = {2025-05-01},
journal = {Plant Direct},
volume = {9},
number = {5},
pages = {e70051},
abstract = {Post-translational modifications of histones (PTMs) play a crucial role in regulating chromatin function. These modifications are integral to numerous biological processes, including transcription, DNA repair, replication, and chromatin remodeling. Although several PTMs have been identified, enhancing our understanding of their roles in these processes, there is still much to discover given the potential for virtually any histone residue to be modified. In this study, we report the discovery of a novel PTM in the model diatom , glutamate methylation identified by mass spectrometry at multiple positions on histone H4 and at position 96 on histone H2B. This modification was also detected in other model organisms, including , , and humans, but not in . Structural bioinformatics analyses, including molecular dynamics simulations, revealed that methylation of glutamate residues on histones induces displacement of these residues, exposing them to solvent and disrupting interactions with neighboring residues in associated histones. This disruption may interfere with histone complexes promoting histone eviction or facilitating interactions with regulatory proteins or complexes, which may compromise the overall nucleosome stability.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Achouba, Yanis; Peres, Basile; Ascoët, Steven; Meudal, Hervé; Caumes, Cécile; Zoukimian, Claude; Millet, Hugo; Choteau‐Bodor, Maureen; Carvalhosa, Cathy; Croyal, Mikael; Bouchama, Fella; Wulff, Heike; Téletchéa, Stéphane; Béroud, Rémy; Ishow, Eléna; Landon, Céline; Boumendjel, Ahcène; Montnach, Jérôme; Waard, Michel De
Photoisomerization of Azobenzene‐Extended Charybdotoxin for the Optical Control of K<sub>v</sub>1.2 Potassium Channel Activity Article de journal
Dans: Angew Chem Int Ed, 2025, ISSN: 1521-3773.
@article{Achouba2025,
title = {Photoisomerization of Azobenzene‐Extended Charybdotoxin for the Optical Control of K_{v}1.2 Potassium Channel Activity},
author = {Yanis Achouba and Basile Peres and Steven Ascoët and Hervé Meudal and Cécile Caumes and Claude Zoukimian and Hugo Millet and Maureen Choteau‐Bodor and Cathy Carvalhosa and Mikael Croyal and Fella Bouchama and Heike Wulff and Stéphane Téletchéa and Rémy Béroud and Eléna Ishow and Céline Landon and Ahcène Boumendjel and Jérôme Montnach and Michel De Waard},
doi = {10.1002/anie.202423278},
issn = {1521-3773},
year = {2025},
date = {2025-03-09},
urldate = {2025-03-09},
journal = {Angew Chem Int Ed},
publisher = {Wiley},
abstract = {<jats:title>Abstract</jats:title><jats:p>Natural peptides from animal venoms effectively modulate ion channel activity. While photoswitches regulate small compound pharmacology, their application to natural peptides rich in disulfide bridges and active on ion channels is novel due to larger pharmacophores. A pilot study integrating azobenzene photoswitches into charybdotoxin (ChTx), known for blocking potassium channels is initiated. Two click‐chemistry‐compatible azobenzene are synthesized differing in length and amide orientation (Az<jats:sub>1</jats:sub> & Az<jats:sub>2</jats:sub>). Az<jats:sub>1</jats:sub> is grafted onto ChTx at various amino acid positions using L‐azidohomoalanine mutation. ChTx monomers outperformed dimers, particularly with azobenzene at position 14, by exhibiting optimal photoswitching activity. In the <jats:italic>cis</jats:italic> configuration, Az<jats:sub>1</jats:sub> altered ChTx's pharmacophore, reducing potassium channel blockage, while conversely, Az<jats:sub>2</jats:sub> increased ChTx potency. This study pioneers photoswitch application to complex peptides, leveraging structure‐activity relationships. Successful integration depends on precise azobenzene positioning and chemical grafting guided by SAR insights. This advancement underscores the adaptability of photoswitch technology to intricate peptide structures, offering new avenues for pharmacological modulation.</jats:p>},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Muñoz-Garcia, Javier; Vargas-Franco, Jorge W.; Schiavone, Kristina; Keatinge, Marcus T.; Young, Robin; Amiaud, Jérôme; Fradet, Laurie; Jégou, Jean-François; Yagita, Hideo; Blin-Wakkach, Claudine; Wakkach, Abdelilah; Cochonneau, Denis; Ollivier, Emilie; Pugière, Martine; Henriquet, Corinne; Legendre, Marie; Giurgea, Irina; Amselem, Serge; Heymann, Marie-Françoise; Télétchéa, Stéphane; Lézot, Frédéric; Heymann, Dominique
Interleukin-34 orchestrates bone formation through its binding to bone morphogenic proteins Article de journal
Dans: Theranostics, vol. 15, no. 7, p. 3185–3202, 2025, ISSN: 1838-7640.
@article{Muñoz-Garcia2025,
title = {Interleukin-34 orchestrates bone formation through its binding to bone morphogenic proteins},
author = {Javier Muñoz-Garcia and Jorge W. Vargas-Franco and Kristina Schiavone and Marcus T. Keatinge and Robin Young and Jérôme Amiaud and Laurie Fradet and Jean-François Jégou and Hideo Yagita and Claudine Blin-Wakkach and Abdelilah Wakkach and Denis Cochonneau and Emilie Ollivier and Martine Pugière and Corinne Henriquet and Marie Legendre and Irina Giurgea and Serge Amselem and Marie-Françoise Heymann and Stéphane Télétchéa and Frédéric Lézot and Dominique Heymann},
url = {https://univ-poitiers.hal.science/hal-04901466/},
doi = {10.7150/thno.107340},
issn = {1838-7640},
year = {2025},
date = {2025-02-11},
urldate = {2025-02-11},
journal = {Theranostics},
volume = {15},
number = {7},
pages = {3185--3202},
publisher = {Ivyspring International Publisher},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
2 publications
Goux, Marine; Demonceaux, Marie; Hendrickx, Johann; Solleux, Claude; Lormeau, Emilie; Fredslund, Folmer; Tezé, David; Offmann, Bernard; André-Miral, Corinne
Sucrose phosphorylase from Alteromonas mediterranea: structural insight into the regioselective α-glucosylation of (+)-catechin Article de journal
Dans: Biochimie, 2024.
@article{Goux2023.04.11.536264,
title = {Sucrose phosphorylase from Alteromonas mediterranea: structural insight into the regioselective α-glucosylation of (+)-catechin},
author = {Marine Goux and Marie Demonceaux and Johann Hendrickx and Claude Solleux and Emilie Lormeau and Folmer Fredslund and David Tezé and Bernard Offmann and Corinne André-Miral},
url = {https://www.biorxiv.org/content/10.1101/2023.04.11.536264v2
hal-04095395v2 },
doi = {10.1016/j.biochi.2024.01.004},
year = {2024},
date = {2024-01-09},
urldate = {2024-01-09},
journal = {Biochimie},
publisher = {Cold Spring Harbor Laboratory},
abstract = {Sucrose phosphorylases, through transglycosylation reactions, are interesting enzymes that can transfer regioselectively glucose from sucrose, the donor substrate, onto acceptors like flavonoids to form glycoconjugates and hence modulate their solubility and bioactivity. Here, we report for the first time the structure of sucrose phosphorylase from the marine bacteria Alteromonas mediterranea (AmSP) and its enzymatic properties. Kinetics of sucrose hydrolysis and transglucosylation capacities on (+)-catechin were investigated. Wild-type enzyme (AmSP-WT) displayed high hydrolytic activity on sucrose and was devoid of transglucosylation activity on (+)-catechin. Two variants, AmSP-Q353F and AmSP-P140D catalysed the regiospecific transglucosylation of (+)-catechin: 89 % of a novel compound (+)-catechin-4′-O-α-d-glucopyranoside (CAT-4′) for AmSP-P140D and 92 % of (+)-catechin-3′-O-α-d-glucopyranoside (CAT-3′) for AmSP-Q353F. The compound CAT-4′ was fully characterized by NMR and mass spectrometry. An explanation for this difference in regiospecificity was provided at atomic level by molecular docking simulations: AmSP-P140D was found to preferentially bind (+)-catechin in a mode that favours glucosylation on its hydroxyl group in position 4′ while the binding mode in AmSP-Q353F favoured glucosylation on its hydroxyl group in position 3’.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Graton, Jérôme; Goupille, Anaïs; Ferré, Tanguy; Offmann, Bernard; André-Miral, Corinne; Questel, Jean-Yves Le
Antioxidant properties of catechin and its 3′O-α-glucoside: Insights from computational chemistry calculations Article de journal
Dans: Computational and Theoretical Chemistry, vol. 1236, p. 114608, 2024, ISSN: 2210-271X.
@article{GRATON2024114608,
title = {Antioxidant properties of catechin and its 3′O-α-glucoside: Insights from computational chemistry calculations},
author = {Jérôme Graton and Anaïs Goupille and Tanguy Ferré and Bernard Offmann and Corinne André-Miral and Jean-Yves Le Questel},
url = {https://www.sciencedirect.com/science/article/pii/S2210271X24001476
https://hal.science/hal-04610796v1},
doi = {https://doi.org/10.1016/j.comptc.2024.114608},
issn = {2210-271X},
year = {2024},
date = {2024-01-01},
urldate = {2024-01-01},
journal = {Computational and Theoretical Chemistry},
volume = {1236},
pages = {114608},
abstract = {Density functional theory (DFT) calculations were used to investigate the conformational landscape of catechin and one of its main glucoside derivative (catechin-3′ O- α −glucopyranoside), and to determine the corresponding antioxidant properties. These investigations were carried out in benzene and water using the SMD universal continuum solvation model. Both properties were found to be significantly affected. The structures are characterized in both solvents by strong intramolecular hydrogen bonds (IMHB). In an apolar environment, Hydrogen Atom Transfer (HAT) is by far favored whereas in water the Sequential Proton Loss Electron Transfer (SPLET) mechanism is strongly preferred. In benzene, the catechin fragment has the best antioxidant character (from 27 kJ/mole) whereas in polar surroundings, the glucoside derivative has a slightly better antiradical activity (from 5 kJ/mole). Our results confirm the key role of the 3′-OH and 4′-OH groups of the catechole ring in these properties.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
7 publications
Venturell, Paul; Teletchea, Stéphane; Bartolozzi P. Bales A.M., Bird A. T.; Teletchea, Fabrice
Dans: Cybium, vol. 47, iss. 3, p. 315-323, 2023, ISBN: 0399-0974.
@article{nokey,
title = {A student-based expansion of the strategies of reproduction in fish (STOREFISH) database to 288 North American freshwater and anadromous species for 14 egg and larval traits},
author = {Paul Venturell and Stéphane Teletchea and Bales A.M., Bartolozzi P., Bird A.T., Blevins T.K., Campaniello S.J., Caizergue M., Carlu L., Chancerelle G., Colletta B., Dauphin L., Doche B., Derolf P.M., De Wever T., Dewig E.M., Dixon L.M., Durand C., Eck M., Faatauira T., Fisher S.M., Fix G., Fournier S., Gauthy A., Golitin C., Guyader S., Hachet F., Harnay P., Hawkins S.G., Kaufling A., Khan M., Kesterson W.J., Klein M.K., Lejeune C., Loiseau J., Loyau R., Luginbuhl S.B., Maeso J., Marc T., Martineau L., Meurillon T., Mesnieres E., Mohra R., Mccord A.O., Mcdonald Z.N., Mckay A.B., Miller T., Minhinnett S.R., Poujoulat R., Profit V., Psurny G.Q., Raymond G., Redinger R.R., Rech G., Rider A.L., Rodriguez L., Sanders S.S., Salou G., Saucier T.E., Schwer J.D., Seymour R.D., Seznec C., Shook B.L., Soler J., Tettling L., Thornburg G.E., Tottoli T., Veber E., Verdier L., Verin R., Vigot M., Vigouroux E., Voss K.N., Weir J.L. and Fabrice Teletchea},
editor = {Société Française d'Ichtyologie},
url = {https://sfi-cybium.fr/fr/student-based-expansion-strategies-reproduction-fish-storefish-database-288-north-american},
doi = {10.26028/cybium/2023-006},
isbn = {0399-0974},
year = {2023},
date = {2023-07-01},
urldate = {2023-07-01},
journal = {Cybium},
volume = {47},
issue = {3},
pages = {315-323},
abstract = {Teleosts exhibit the highest reproductive diversity of all vertebrates, but this diversity has not been extensively analyzed, in part due to a lack of synthesis of life history information. The original STOREFISH (STrategies Of REproduction in FISH) database was published in 2007, and then released online in 2020 to facilitate data visualization and utilization (www.storefish.org). The original database contains information on 50 life history traits from ~1,200 references for 80 freshwater and anadromous species, mostly from Europe. Here, we describe the process and results of an international effort to update and extend the database for 14 egg and larval traits from North American freshwater and anadromous species, and then reassess previous bivari- ate relationships. Students in the United States and France used data from nearly 800 references to increase the STOREFISH database to 8,081 records (70% increase) for 368 species (360% increase) and 41 families (116% increase). We extracted fewer records per species than the original database because we included many species for which relatively little information was known. However, the distribution of records among trait values was similar to the original database. Updating and expanding the database improved the accuracy of the incuba- tion time-temperature relationship below 10°C, and challenged a previous assumption regarding the larval size- egg diameter relationship. Our expansion effort progressed smoothly and quickly via an educational model that emphasized supervised research and collaboration. We are extending this approach to include validators for data curation, and both pure and applied research that demonstrates the utility of the STOREFISH database to biodi- versity research, conservation, assessment, management, and aquaculture.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Sanejouand, Yves-Henri
On the unknown proteins of eukaryotic proteomes Article de journal
Dans: Journal of Molecular Evolution, vol. 91, p. 492-501, 2023.
@article{sanejouand2023unknown,
title = {On the unknown proteins of eukaryotic proteomes},
author = {Yves-Henri Sanejouand},
url = {https://doi.org/10.1007/s00239-023-10116-1
hal-03863835},
doi = {10.1007/s00239-023-10116-1},
year = {2023},
date = {2023-05-23},
urldate = {2023-05-23},
journal = {Journal of Molecular Evolution},
volume = {91},
pages = {492-501},
abstract = {In order to study unknown proteins on a large scale, a reference system has been set up for the three major eukaryotic lineages, built with 36 proteomes as taxonomically diverse as possible. Proteins from 362 eukaryotic proteomes with no known homologue in this set were then analyzed, focusing noteworthy on singletons, that is, on unknown proteins with no known homologue in their own proteome. Consistently, according to Uniprot, for a given species, no more than 12% of the singletons thus found are known at the protein level. Also, since they rely on the information found in the alignment of homologous sequences, predictions of AlphaFold2 for their tridimensional structure are usually poor. In the case of metazoan species, the number of singletons seems to increase as a function of the evolutionary distance from the reference system. Interestingly, no such trend is found in the cases of viridiplantae and fungi, as if the timescale on which singletons are added to proteomes were different in metazoa and in other eukaryotic kingdoms. In order to confirm this phenomenon, further studies of proteomes closer to those of the reference system are however needed.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Demonceaux, Marie; Goux, Marine; Schimith, Lucia Emanueli; Santos, Michele Goulart Dos; Hendrickx, Johann; Offmann, Bernard; André-Miral, Corinne
Enzymatic synthesis, characterization and molecular docking of a new functionalized polyphenol: Resveratrol-3, 4’-⍺-diglucoside Article de journal
Dans: Results in Chemistry, p. 100956, 2023.
@article{demonceaux2023enzymatic,
title = {Enzymatic synthesis, characterization and molecular docking of a new functionalized polyphenol: Resveratrol-3, 4’-⍺-diglucoside},
author = {Marie Demonceaux and Marine Goux and Lucia Emanueli Schimith and Michele Goulart Dos Santos and Johann Hendrickx and Bernard Offmann and Corinne André-Miral},
url = {https://www.sciencedirect.com/science/article/pii/S2211715623001959},
doi = {10.1016/j.rechem.2023.100956},
year = {2023},
date = {2023-05-16},
urldate = {2023-05-16},
journal = {Results in Chemistry},
pages = {100956},
publisher = {Elsevier},
abstract = {Transglucosylation of resveratrol by the Q345F variant of sucrose phosphorylase from Bifidobacterium adolescentis (BaSP) was extensively studied during the last decade. Indeed, Q345F is able to catalyze the synthesis of resveratrol-3-O-⍺-D-glucoside (RES-3) with yield up to 97% using a cost-effective glucosyl donor, sucrose (Kraus et al., Chemical Communications, 53(90), 12182–12184 (2017)). Despite the fact that two further products were detectable in low amounts after glucoside synthesis, they were never identified. Here, we isolated and fully characterized one of those two minor products: resveratrol-3,4′-O-⍺-D-diglucoside (RES-3,4′). This original compound had never been described before. Using bioinformatics models, we successfully explained the formation of this diglucosylated product. Indeed, with RES-3 as acceptor substrate, Q345F is able to transfer a glucosyl moiety in position 4′-OH, what had been reported as impossible in the literature. The low yield observed is due to the steric hindrance into the catalytic site between RES-3 and residues Tyr132 and Tyr344. Nevertheless, the substrate orientation in the active site is favored by stabilizing interactions. Ring A of RES-3 bearing the diol moiety is stabilized by hydrogen bonds with residues Asp50, Arg135, Asn347 and Arg399. Hydroxyl group OH-4′ shares hydrogen bonds with the catalytic residues Asp192 and Glu232. Multiple hydrophobic contacts complete the stabilization of the substrate to favor the glucosylation at position 4′. Understanding of the mechanisms allowing the glucosylation at position 4′ of resveratrol will help the development of enzymatic tools to target and control the enzymatic synthesis of original ⍺-glucosylated polyphenols with high added value and better biodisponibility.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Téletchéa, Stéphane; Esque, Jérémy; Urbain, Aurélie; Etchebest, Catherine; de Brevern, Alexandre G.
Evaluation of Transmembrane Protein Structural Models Using HPMScore Article de journal
Dans: BioMedInformatics, vol. 3, no. 2, p. 306–326, 2023, ISSN: 2673-7426.
@article{biomedinformatics3020021,
title = {Evaluation of Transmembrane Protein Structural Models Using HPMScore},
author = {Stéphane Téletchéa and Jérémy Esque and Aurélie Urbain and Catherine Etchebest and Alexandre G. de Brevern},
url = {https://www.mdpi.com/2673-7426/3/2/21
https://hal.science/hal-03251546v1, HAL},
doi = {10.3390/biomedinformatics3020021},
issn = {2673-7426},
year = {2023},
date = {2023-05-02},
urldate = {2023-05-02},
journal = {BioMedInformatics},
volume = {3},
number = {2},
pages = {306--326},
abstract = {Transmembrane proteins (TMPs) are a class of essential proteins for biological and therapeutic purposes. Despite an increasing number of structures, the gap with the number of available sequences remains impressive. The choice of a dedicated function to select the most probable/relevant model among hundreds is a specific problem of TMPs. Indeed, the majority of approaches are mostly focused on globular proteins. We developed an alternative methodology to evaluate the quality of TMP structural models. HPMScore took into account sequence and local structural information using the unsupervised learning approach called hybrid protein model. The methodology was extensively evaluated on very different TMP all-α proteins. Structural models with different qualities were generated, from good to bad quality. HPMScore performed better than DOPE in recognizing good comparative models over more degenerated models, with a Top 1 of 46.9% against DOPE 40.1%, both giving the same result in 13.0%. When the alignments used are higher than 35%, HPM is the best for 52%, against 36% for DOPE (12% for both). These encouraging results need further improvement particularly when the sequence identity falls below 35%. An area of enhancement would be to train on a larger training set. A dedicated web server has been implemented and provided to the scientific community. It can be used with structural models generated from comparative modeling to deep learning approaches.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Demonceaux, Marie; Goux, Marine; Hendrickx, Johann; Solleux, Claude; Cadet, Frédéric; Lormeau, Émilie; Offmann, Bernard; André-Miral, Corinne
Regioselective glucosylation of (+)-catechin using a new variant of sucrose phosphorylase from Bifidobacterium adolescentis Article de journal
Dans: Organic & Biomolecular Chemistry, vol. 21, no. 11, p. 2307–2311, 2023.
@article{demonceaux2023regioselective,
title = {Regioselective glucosylation of (+)-catechin using a new variant of sucrose phosphorylase from Bifidobacterium adolescentis},
author = {Marie Demonceaux and Marine Goux and Johann Hendrickx and Claude Solleux and Frédéric Cadet and Émilie Lormeau and Bernard Offmann and Corinne André-Miral},
doi = {10.1039/D3OB00191A},
year = {2023},
date = {2023-02-22},
urldate = {2023-02-22},
journal = {Organic & Biomolecular Chemistry},
volume = {21},
number = {11},
pages = {2307--2311},
publisher = {Royal Society of Chemistry},
abstract = {Mutation Q345F in sucrose phosphorylase from Bifidobacterium adolescentis (BaSP) has shown to allow efficient (+)-catechin glucosylation yielding a regioisomeric mixture: (+)-catechin-3′-O-α-D-glucopyranoside, (+)-catechin-5-O-α-D-glucopyranoside and (+)-catechin-3′,5-O-α-D-diglucopyranoside with a ratio of 51 : 25 : 24. Here, we efficiently increased the control of (+)-catechin glucosylation regioselectivity with a new variant Q345F/P134D. The same products were obtained with a ratio of 82 : 9 : 9. Thanks to bioinformatics models, we successfully explained the glucosylation favoured at the OH-3′ position due to the mutation P134D.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Poulet, Axel; Rousselot, Ellyn; Téletchéa, Stéphane; Noirot, Céline; Jacob, Yannick; Wolfswinkel, Josien; Thiriet, Christophe; Duc, Céline
The Histone Chaperone Network Is Highly Conserved in Physarum polycephalum Article de journal
Dans: International Journal of Molecular Sciences, vol. 24, no. 2, 2023, ISSN: 1422-0067.
@article{ijms24021051,
title = {The Histone Chaperone Network Is Highly Conserved in Physarum polycephalum},
author = {Axel Poulet and Ellyn Rousselot and Stéphane Téletchéa and Céline Noirot and Yannick Jacob and Josien Wolfswinkel and Christophe Thiriet and Céline Duc},
url = {https://www.mdpi.com/1422-0067/24/2/1051
hal-03978828v1 },
doi = {10.3390/ijms24021051},
issn = {1422-0067},
year = {2023},
date = {2023-01-01},
urldate = {2023-01-01},
journal = {International Journal of Molecular Sciences},
volume = {24},
number = {2},
abstract = {The nucleosome is composed of histones and DNA. Prior to their deposition on chromatin, histones are shielded by specialized and diverse proteins known as histone chaperones. They escort histones during their entire cellular life and ensure their proper incorporation in chromatin. Physarum polycephalum is a Mycetozoan, a clade located at the crown of the eukaryotic tree. We previously found that histones, which are highly conserved between plants and animals, are also highly conserved in Physarum. However, histone chaperones differ significantly between animal and plant kingdoms, and this thus probed us to further study the conservation of histone chaperones in Physarum and their evolution relative to animal and plants. Most of the known histone chaperones and their functional domains are conserved as well as key residues required for histone and chaperone interactions. Physarum is divergent from yeast, plants and animals, but PpHIRA, PpCABIN1 and PpSPT6 are similar in structure to plant orthologues. PpFACT is closely related to the yeast complex, and the Physarum genome encodes the animal-specific APFL chaperone. Furthermore, we performed RNA sequencing to monitor chaperone expression during the cell cycle and uncovered two distinct patterns during S-phase. In summary, our study demonstrates the conserved role of histone chaperones in handling histones in an early-branching eukaryote.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Mam, Bhavika; Tsitsanou, Katerina E.; Liggri, Panagiota G. V.; Saitta, Francesca; Stamati, Evgenia C. V.; Mahita, Jarjapu; Leonis, Georgios; Drakou, Christina E.; Papadopoulos, Manthos; Arnaud, Philippe; Offmann, Bernard; Fessas, Dimitrios; Sowdhamini, Ramanathan; Zographos, Spyros E.
Influence of pH on indole-dependent heterodimeric interactions between Anopheles gambiae odorant-binding proteins OBP1 and OBP4 Article de journal
Dans: International Journal of Biological Macromolecules, vol. 245, p. 125422, 2023, ISSN: 0141-8130.
@article{MAM2023125422,
title = {Influence of pH on indole-dependent heterodimeric interactions between Anopheles gambiae odorant-binding proteins OBP1 and OBP4},
author = {Bhavika Mam and Katerina E. Tsitsanou and Panagiota G. V. Liggri and Francesca Saitta and Evgenia C. V. Stamati and Jarjapu Mahita and Georgios Leonis and Christina E. Drakou and Manthos Papadopoulos and Philippe Arnaud and Bernard Offmann and Dimitrios Fessas and Ramanathan Sowdhamini and Spyros E. Zographos},
url = {https://www.sciencedirect.com/science/article/pii/S0141813023023164},
doi = {https://doi.org/10.1016/j.ijbiomac.2023.125422},
issn = {0141-8130},
year = {2023},
date = {2023-01-01},
urldate = {2023-01-01},
journal = {International Journal of Biological Macromolecules},
volume = {245},
pages = {125422},
abstract = {Insect Odorant Binding Proteins (OBPs) constitute important components of their olfactory apparatus, as they are essential for odor recognition. OBPs undergo conformational changes upon pH change, altering their interactions with odorants. Moreover, they can form heterodimers with novel binding characteristics. Anopheles gambiae OBP1 and OBP4 were found capable of forming heterodimers possibly involved in the specific perception of the attractant indole. In order to understand how these OBPs interact in the presence of indole and to investigate the likelihood of a pH-dependent heterodimerization mechanism, the crystal structures of OBP4 at pH 4.6 and 8.5 were determined. Structural comparison to each other and with the OBP4-indole complex (3Q8I, pH 6.85) revealed a flexible N-terminus and conformational changes in the α4-loop-α5 region at acidic pH. Fluorescence competition assays showed a weak binding of indole to OBP4 that becomes further impaired at acidic pH. Additional Molecular Dynamic and Differential Scanning Calorimetry studies displayed that the influence of pH on OBP4 stability is significant compared to the modest effect of indole. Furthermore, OBP1-OBP4 heterodimeric models were generated at pH 4.5, 6.5, and 8.5, and compared concerning their interface energy and cross-correlated motions in the absence and presence of indole. The results indicate that the increase in pH may induce the stabilization of OBP4 by increasing its helicity, thereby enabling indole binding at neutral pH that further stabilizes the protein and possibly promotes the creation of a binding site for OBP1. A decrease in interface stability and loss of correlated motions upon transition to acidic pH may provoke the heterodimeric dissociation allowing indole release. Finally, we propose a potential OBP1-OBP4 heterodimer formation/disruption mechanism induced by pH change and indole binding.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
1 publication
Sanejouand, Yves-Henri
At least three xenon binding sites in the glycine binding domain of the N-methyl D-aspartate receptor Article de journal
Dans: Archives of biochemistry and biophysics, vol. 724, p. 109265, 2022, (arXiv: 2203.02219).
@article{sanejouand_at_2022,
title = {At least three xenon binding sites in the glycine binding domain of the N-methyl D-aspartate receptor},
author = {Yves-Henri Sanejouand},
url = {http://arxiv.org/abs/2203.02219
hal-03863820v1 },
doi = {https://doi.org/10.1016/j.abb.2022.109265},
year = {2022},
date = {2022-03-01},
urldate = {2022-03-01},
journal = {Archives of biochemistry and biophysics},
volume = {724},
pages = {109265},
abstract = {Xenon can produce general anesthesia. Its main protein target is the N-methyl-D-aspartate receptor, a ionotropic channel playing a pivotal role in the function of the central nervous system. The molecular mechanisms allowing this noble gas to have such a specific effect remain obscure, probably as a consequence of the lack of structural data at the atomic level of detail. Herein, as a result of five independent molecular dynamics simulations, three different binding sites were found for xenon in the glycine binding domain of the Nmethyl-D-aspartate receptor. The absolute binding free energy of xenon in these sites ranges between -8 and -14 kJ·mole−1. However, it depends significantly upon the protein conformer chosen for performing the calculation, suggesting that larger values could probably be obtained, if other conformers were considered. These three sites are next to each other, one of them being next to the glycine site. This could explain why the F758W and F758Y mutations can prevent competitive inhibition by xenon without affecting glycine binding.},
note = {arXiv: 2203.02219},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
8 publications
Guo, Xia; Xuan, Ning; Liu, Guoxia; Xie, Hongyan; Lou, Qinian; Arnaud, Philippe; Offmann, Bernard; Picimbon, Jean-François
An Expanded Survey of the Moth PBP/GOBP Clade in Bombyx mori: New Insight into Expression and Functional Roles Article de journal
Dans: Frontiers in Physiology, vol. 12, p. 1701, 2021, ISSN: 1664-042X.
@article{10.3389/fphys.2021.712593,
title = {An Expanded Survey of the Moth PBP/GOBP Clade in Bombyx mori: New Insight into Expression and Functional Roles},
author = {Xia Guo and Ning Xuan and Guoxia Liu and Hongyan Xie and Qinian Lou and Philippe Arnaud and Bernard Offmann and Jean-François Picimbon},
url = {https://www.frontiersin.org/article/10.3389/fphys.2021.712593},
doi = {10.3389/fphys.2021.712593},
issn = {1664-042X},
year = {2021},
date = {2021-10-28},
urldate = {2021-01-01},
journal = {Frontiers in Physiology},
volume = {12},
pages = {1701},
abstract = {We studied the expression profile and ontogeny (from the egg stage through the larval stages and pupal stages, to the elderly adult age) of four OBPs from the silkworm moth Bombyx mori. We first showed that male responsiveness to female sex pheromone in the silkworm moth B. mori does not depend on age variation; whereas the expression of BmorPBP1, BmorPBP2, BmorGOBP1, and BmorGOBP2 varies with age. The expression profile analysis revealed that the studied OBPs are expressed in non-olfactory tissues at different developmental stages. In addition, we tested the effect of insecticide exposure on the expression of the four OBPs studied. Exposure to a toxic macrolide insecticide endectocide molecule (abamectin) led to the modulated expression of all four genes in different tissues. The higher expression of OBPs was detected in metabolic tissues, such as the thorax, gut, and fat body. All these data strongly suggest some alternative functions for these proteins other than olfaction. Finally, we carried out ligand docking studies and reported that PBP1 and GOBP2 have the capacity of binding vitamin K1 and multiple different vitamins.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Teze, David; Zhao, Jiao; Wiemann, Mathias; Ara, Kazi Z G; Lupo, Rossana; Zeuner, Birgitte; Vuillemin, Marlène; Rønne, Mette E; Carlström, Göran; Duus, Jens Ø; Sanejouand, Yves-Henri; O'Donohue, Michael J; Karlsson, Eva Nordberg; Fauré, Régis; Stålbrand, Henrik; Svensson, Birte
Rational Enzyme Design without Structural Knowledge: A Sequence-Based Approach for Efficient Generation of Transglycosylases Article de journal
Dans: Chemistry – A European Journal, vol. 27, no. 40, p. 10323–10334, 2021, ISSN: 0947-6539, 1521-3765.
@article{https://doi.org/10.1002/chem.202100110,
title = {Rational Enzyme Design without Structural Knowledge: A Sequence-Based Approach for Efficient Generation of Transglycosylases},
author = {David Teze and Jiao Zhao and Mathias Wiemann and Kazi Z G Ara and Rossana Lupo and Birgitte Zeuner and Marlène Vuillemin and Mette E Rønne and Göran Carlström and Jens Ø Duus and Yves-Henri Sanejouand and Michael J O'Donohue and Eva Nordberg Karlsson and Régis Fauré and Henrik Stålbrand and Birte Svensson},
url = {https://chemistry-europe.onlinelibrary.wiley.com/doi/abs/10.1002/chem.202100110},
doi = {https://doi.org/10.1002/chem.202100110},
issn = {0947-6539, 1521-3765},
year = {2021},
date = {2021-04-29},
urldate = {2021-04-29},
journal = {Chemistry – A European Journal},
volume = {27},
number = {40},
pages = {10323--10334},
abstract = {Glycobiology is dogged by the relative scarcity of synthetic, defined oligosaccharides. Enzyme-catalysed glycosylation using glycoside hydrolases is feasible but is hampered by the innate hydrolytic activity of these enzymes. Protein engineering is useful to remedy this, but it usually requires prior structural knowledge of the target enzyme, and/or relies on extensive, time-consuming screening and analysis. Here we describe a straightforward strategy that involves rational rapid in silico analysis of protein sequences. The method pinpoints 6‒12 single mutant candidates to improve transglycosylation yields. Requiring very little prior knowledge of the target enzyme other than its sequence, the method is generic and procures catalysts for the formation of glycosidic bonds involving various d / l -, α/β-pyranosides or furanosides, and exo - and endo -action. Moreover, mutations validated in one enzyme can be transposed to others, even distantly related enzymes.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Sanejouand, Yves-Henri
Normal-mode driven exploration of protein domain motions Article de journal
Dans: J. Comput. Chem., vol. 42, p. 2250, 2021.
@article{sanejouand2021normalmode,
title = {Normal-mode driven exploration of protein domain motions},
author = {Yves-Henri Sanejouand},
url = {https://arxiv.org/abs/2103.11959},
doi = {10.1002/jcc.26755},
year = {2021},
date = {2021-03-22},
urldate = {2021-03-22},
journal = {J. Comput. Chem.},
volume = {42},
pages = {2250},
abstract = {Domain motions involved in the function of proteins can often be well described as a combination of motions along a handfull of low-frequency modes, that is, with the values of a few normal coordinates. This means that, when the functional motion of a protein is unknown, it should prove possible to predict it, since it amounts to guess a few values. However, without the help of additional experimental data, using normal coordinates for generating accurate conformers far away from the initial one is not so straightforward. To do so, a new approach is proposed: instead of building conformers directly with the values of a subset of normal coordinates, they are built in two steps, the conformer built with normal coordinates being just used for defining a set of distance constraints, the final conformer being built so as to match them. Note that this approach amounts to transform the problem of generating accurate protein conformers using normal coordinates into a better known one: the distance-geometry problem, which is herein solved with the help of the ROSETTA software. In the present study, this approach allowed to rebuild accurately six large amplitude conformational changes, using at most six low-frequency normal coordinates. As a consequence of the low-dimensionality of the corresponding subspace, random exploration also proved enough for generating low-energy conformers close to the known end-point of the conformational change of the LAO binding protein, lysozyme T4 and adenylate kinase.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Sanejouand, Yves-Henri
On the vibrational free energy of hydrated proteins Article de journal
Dans: Physical Biology, vol. 18, no. 3, p. 036003, 2021.
@article{Sanejouand_2021,
title = {On the vibrational free energy of hydrated proteins},
author = {Yves-Henri Sanejouand},
url = {https://doi.org/10.1088/1478-3975/abdc0f},
doi = {10.1088/1478-3975/abdc0f},
year = {2021},
date = {2021-03-01},
urldate = {2021-03-01},
journal = {Physical Biology},
volume = {18},
number = {3},
pages = {036003},
publisher = {IOP Publishing},
abstract = {When the hydration shell of a protein is filled with at least 0.6 gram of water per gram of protein, a significant anti-correlation between the vibrational free energy and the potential energy of energy-minimized conformers is observed. This means that low potential energy, well-hydrated, protein conformers tend to be more rigid than high-energy ones. On the other hand, in the case of CASP target 624, when its hydration shell is filled, a significant energy gap is observed between the crystal structure and the best conformers proposed during the prediction experiment, strongly suggesting that including explicit water molecules may help identifying unlikely conformers among good-looking ones.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Floch, Aline; Téletchéa, Stéphane; Tournamille, Christophe; de Brevern, Alexandre G; Pirenne, France
A Review of the Literature Organized Into a New Database: RHeference Article de journal
Dans: Transfusion Medicine Reviews, 2021, ISSN: 0887-7963.
@article{FLOCH2021,
title = {A Review of the Literature Organized Into a New Database: RHeference},
author = {Aline Floch and Stéphane Téletchéa and Christophe Tournamille and Alexandre G de Brevern and France Pirenne},
url = {https://www.sciencedirect.com/science/article/pii/S0887796321000109},
doi = {https://doi.org/10.1016/j.tmrv.2021.04.002},
issn = {0887-7963},
year = {2021},
date = {2021-01-01},
journal = {Transfusion Medicine Reviews},
abstract = {Hundreds of articles containing heterogeneous data describe D variants or add to the knowledge of known alleles. Data can be difficult to find despite existing online blood group resources and genetic and literature databases. We have developed a modern, elaborate database for D variants, thanks to an extensive literature search with meticulous curation of 387 peer-reviewed articles and 80 abstracts from major conferences and other sources. RHeference contains entries for 710 RHD alleles, 11 RHCE alleles, 30 phenotype descriptions (preventing data loss from historical sources), 35 partly characterized alleles, 3 haplotypes, and 16 miscellaneous entries. The entries include molecular, phenotypic, serological, alloimmunization, haplotype, geographical, and other data, detailed for each source. The main characteristics are summarized for each entry. The sources for all information are included and easily accessible through doi and PMID links. Overall, the database contains more than 10,000 individual pieces of data. We have set up the database architecture based on our previous expertise on database setup and biocuration for other topics, using modern technologies such as the Django framework, BioPython, Bootstrap, and Jquery. This architecture allows an easy access to data and enables simple and complex queries: combining multiple mutations, keywords, or any of the characteristics included in the database. RHeference provides a complement to existing resources and will continue to grow as our knowledge expands and new articles are published. The database url is http://www.rheference.org/.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Gheyouche, Ennys; Bagueneau, Matthias; Loirand, Gervaise; Offmann, Bernard; Téletchéa, Stéphane
Structural Design and Analysis of the RHOA-ARHGEF1 Binding Mode: Challenges and Applications for Protein-Protein Interface Prediction Article de journal
Dans: Frontiers in Molecular Biosciences, vol. 8, p. 643728, 2021, ISSN: 2296-889X.
@article{gheyouche_structural_2021,
title = {Structural Design and Analysis of the RHOA-ARHGEF1 Binding Mode: Challenges and Applications for Protein-Protein Interface Prediction},
author = {Ennys Gheyouche and Matthias Bagueneau and Gervaise Loirand and Bernard Offmann and Stéphane Téletchéa},
doi = {10.3389/fmolb.2021.643728},
issn = {2296-889X},
year = {2021},
date = {2021-01-01},
journal = {Frontiers in Molecular Biosciences},
volume = {8},
pages = {643728},
abstract = {The interaction between two proteins may involve local movements, such as small side-chains re-positioning or more global allosteric movements, such as domain rearrangement. We studied how one can build a precise and detailed protein-protein interface using existing protein-protein docking methods, and how it can be possible to enhance the initial structures using molecular dynamics simulations and data-driven human inspection. We present how this strategy was applied to the modeling of RHOA-ARHGEF1 interaction using similar complexes of RHOA bound to other members of the Rho guanine nucleotide exchange factor family for comparative assessment. In parallel, a more crude approach based on structural superimposition and molecular replacement was also assessed. Both models were then successfully refined using molecular dynamics simulations leading to protein structures where the major data from scientific literature could be recovered. We expect that the detailed strategy used in this work will prove useful for other protein-protein interface design. The RHOA-ARHGEF1 interface modeled here will be extremely useful for the design of inhibitors targeting this protein-protein interaction (PPI).},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Poulet, Axel; Mishra, Laxmi Narayan; Téletchéa, Stéphane; Hayes, Jeffrey J; Jacob, Yannick; Thiriet, Christophe; Duc, Céline
Identification and characterization of histones in Physarum polycephalum evidence a phylogenetic vicinity of Mycetozoans to the animal kingdom Article de journal
Dans: NAR Genomics and Bioinformatics, vol. 3, no. 4, 2021, ISSN: 2631-9268, (lqab107).
@article{10.1093/nargab/lqab107,
title = {Identification and characterization of histones in Physarum polycephalum evidence a phylogenetic vicinity of Mycetozoans to the animal kingdom},
author = {Axel Poulet and Laxmi Narayan Mishra and Stéphane Téletchéa and Jeffrey J Hayes and Yannick Jacob and Christophe Thiriet and Céline Duc},
url = {https://doi.org/10.1093/nargab/lqab107
hal-03595485v1 },
doi = {10.1093/nargab/lqab107},
issn = {2631-9268},
year = {2021},
date = {2021-01-01},
urldate = {2021-01-01},
journal = {NAR Genomics and Bioinformatics},
volume = {3},
number = {4},
abstract = {Physarum polycephalum belongs to Mycetozoans, a phylogenetic clade apart from the animal, plant and fungus kingdoms. Histones are nuclear proteins involved in genome organization and regulation and are among the most evolutionary conserved proteins within eukaryotes. Therefore, this raises the question of their conservation in Physarum and the position of this organism within the eukaryotic phylogenic tree based on histone sequences. We carried out a comprehensive study of histones in Physarum polycephalum using genomic, transcriptomic and molecular data. Our results allowed to identify the different isoforms of the core histones H2A, H2B, H3 and H4 which exhibit strong conservation of amino acid residues previously identified as subject to post-translational modifications. Furthermore, we also identified the linker histone H1, the most divergent histone, and characterized a large number of its PTMs by mass spectrometry. We also performed an in-depth investigation of histone genes and transcript structures. Histone proteins are highly conserved in Physarum and their characterization will contribute to a better understanding of the polyphyletic Mycetozoan group. Our data reinforce that P. polycephalum is evolutionary closer to animals than plants and located at the crown of the eukaryotic tree. Our study provides new insights in the evolutionary history of Physarum and eukaryote lineages.},
note = {lqab107},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Muñoz-Garcia, Javier; Cochonneau, Denis; Télétchéa, Stéphane; Moranton, Emilie; Lanoe, Didier; Brion, Régis; Lézot, Frédéric; Heymann, Marie-Françoise; Heymann, Dominique
The twin cytokines interleukin-34 and CSF-1: masterful conductors of macrophage homeostasis Article de journal
Dans: Theranostics, vol. 11, no. 4, p. 1568–1593, 2021, ISSN: 1838-7640.
@article{pmid33408768,
title = {The twin cytokines interleukin-34 and CSF-1: masterful conductors of macrophage homeostasis},
author = {Javier Muñoz-Garcia and Denis Cochonneau and Stéphane Télétchéa and Emilie Moranton and Didier Lanoe and Régis Brion and Frédéric Lézot and Marie-Françoise Heymann and Dominique Heymann},
doi = {10.7150/thno.50683},
issn = {1838-7640},
year = {2021},
date = {2021-01-01},
urldate = {2021-01-01},
journal = {Theranostics},
volume = {11},
number = {4},
pages = {1568--1593},
abstract = {Macrophages are specialized cells that control tissue homeostasis. They include non-resident and tissue-resident macrophage populations which are characterized by the expression of particular cell surface markers and the secretion of molecules with a wide range of biological functions. The differentiation and polarization of macrophages relies on specific growth factors and their receptors. Macrophage-colony stimulating factor (CSF-1) and interleukine-34 (IL-34), also known as "twin" cytokines, are part of this regluatory landscape. CSF-1 and IL-34 share a common receptor, the macrophage-colony stimulating factor receptor (CSF-1R), which is activated in a similar way by both factors and turns on identical signaling pathways. However, there is some discrete differential activation leading to specific activities. In this review, we disscuss recent progress in understanding of the role of the twin cytokines in macrophage differentiation, from their interaction with CSF-1R and the activation of signaling pathways, to their implication in macrophage polarization of non-resident and tissue-resident macrophages. A special focus on IL-34, its involvement in pathophsyiological contexts, and its potential as a theranostic target for macrophage therapy will be proposed.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
8 publications
Dhingra, Surbhi; Sowdhamini, Ramanathan; Sanejouand, Yves-Henri; Cadet, Frédéric; Offmann, Bernard
Customised fragment libraries for ab initio protein structure prediction using a structural alphabet Article de journal
Dans: arXiv:2005.01696, 2020.
@article{Dhingra2020,
title = {Customised fragment libraries for ab initio protein structure prediction using a structural alphabet},
author = {Surbhi Dhingra and Ramanathan Sowdhamini and Yves-Henri Sanejouand and Frédéric Cadet and Bernard Offmann},
url = {https://arxiv.org/pdf/2005.01696.pdf},
year = {2020},
date = {2020-05-01},
journal = {arXiv:2005.01696},
abstract = {Motivation: Computational protein structure prediction has taken over the structural community in past few decades, mostly focusing on the development of Template-Free modelling (TFM) or ab initio modelling protocols. Fragment-based assembly (FBA), falls under this category and is by far the most popular approach to solve the spatial arrangements of proteins. FBA approaches usually rely on sequence based profile comparison to generate fragments from a representative structural database. Here we report the use of Protein Blocks (PBs), a structural alphabet (SA) to perform such sequence comparison and to build customised fragment libraries for TFM. Results: We demonstrate that predicted PB sequences for a query protein can be used to search for high quality fragments that overall cover above 90% of the query. The fragments generated are of minimum length of 11 residues, and fragments that cover more than 30% of the query length were often obtained. Our work shows that PBs can serve as a good way to extract structurally similar fragments from a database of representatives of non-homologous structures and of the proteins that contain less ordered regions.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Ostafe, Raluca; Fontaine, Nicolas; Frank, David; Chong, Matthieu Ng Fuk; Prodanovic, Radivoje; Pandjaitan, Rudy; Offmann, Bernard; Cadet, Frédéric; Fischer, Rainer
One-shot optimization of multiple enzyme parameters: Tailoring glucose oxidase for pH and electron mediators Article de journal
Dans: Biotechnology and Bioengineering, vol. 117, no. 1, p. 17–29, 2020, ISSN: 10970290.
@article{Ostafe2020,
title = {One-shot optimization of multiple enzyme parameters: Tailoring glucose oxidase for pH and electron mediators},
author = {Raluca Ostafe and Nicolas Fontaine and David Frank and Matthieu {Ng Fuk Chong} and Radivoje Prodanovic and Rudy Pandjaitan and Bernard Offmann and Frédéric Cadet and Rainer Fischer},
doi = {10.1002/bit.27169},
issn = {10970290},
year = {2020},
date = {2020-01-01},
journal = {Biotechnology and Bioengineering},
volume = {117},
number = {1},
pages = {17--29},
abstract = {Enzymes are biological catalysts with many industrial applications, but natural enzymes are usually unsuitable for industrial processes because they are not optimized for the process conditions. The properties of enzymes can be improved by directed evolution, which involves multiple rounds of mutagenesis and screening. By using mathematical models to predict the structure–activity relationship of an enzyme, and by defining the optimal combination of mutations in silico, we can significantly reduce the number of bench experiments needed, and hence the time and investment required to develop an optimized product. Here, we applied our innovative sequence–activity relationship methodology (innov'SAR) to improve glucose oxidase activity in the presence of different mediators across a range of pH values. Using this machine learning approach, a predictive model was developed and the optimal combination of mutations was determined, leading to a glucose oxidase mutant (P1) with greater specificity for the mediators ferrocene–methanol (12-fold) and nitrosoaniline (8-fold), compared to the wild-type enzyme, and better performance in three pH-adjusted buffers. The kcat/KM ratio of P1 increased by up to 121 folds compared to the wild type enzyme at pH 5.5 in the presence of ferrocene methanol.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Hendrickx, Johann; Tran, Vinh; Sanejouand, Yves-Henri
Numerous severely twisted N-acetylglucosamine conformations found in the protein databank Article de journal
Dans: Proteins: Structure, Function and Bioinformatics, vol. 88, no. 10, p. 1376–1383, 2020, ISSN: 10970134.
@article{Hendrickx2020,
title = {Numerous severely twisted N-acetylglucosamine conformations found in the protein databank},
author = {Johann Hendrickx and Vinh Tran and Yves-Henri Sanejouand},
doi = {10.1002/prot.25957},
issn = {10970134},
year = {2020},
date = {2020-01-01},
journal = {Proteins: Structure, Function and Bioinformatics},
volume = {88},
number = {10},
pages = {1376--1383},
abstract = {Taking advantage of the known planarity of the N-acetyl group of N-acetylglucosamine, an analysis of the quality of carbohydrate structures found in the protein databank was performed. Few obvious defects of the local geometry of the carbonyl group were observed. However, the N-acetyl group was often found in the less favorable cis conformation (12% of the cases). It was also found severely twisted in numerous instances, especially in structures with a resolution poorer than 1.9 Å determined between 2000 and 2015. Though the automated PDB-REDO procedure has proved able to improve nearly 85% of the structural models deposited to the PDB, and does prove able to cure most severely twisted conformations of the N-acetyl group, it fails to correct its high rate of cis conformations. More generally, for structures with a resolution poorer than 1.6 Å, it produces N-acetylglucosamine models in slightly poorer agreement with experimental data, as measured using real-space correlation coefficients. Significant improvements are thus still needed, at least as far as this carbohydrate structure is concerned.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Dhingra, Surbhi; Sowdhamini, Ramanathan; Cadet, Frédéric; Offmann, Bernard
A glance into the evolution of template-free protein structure prediction methodologies Article de journal
Dans: Biochimie, vol. 175, p. 85 - 92, 2020, ISSN: 0300-9084.
@article{DHINGRA202085,
title = {A glance into the evolution of template-free protein structure prediction methodologies},
author = {Surbhi Dhingra and Ramanathan Sowdhamini and Frédéric Cadet and Bernard Offmann},
url = {http://www.sciencedirect.com/science/article/pii/S0300908420300961},
doi = {https://doi.org/10.1016/j.biochi.2020.04.026},
issn = {0300-9084},
year = {2020},
date = {2020-01-01},
journal = {Biochimie},
volume = {175},
pages = {85 - 92},
abstract = {Prediction of protein structures using computational approaches has been explored for over two decades, paving a way for more focused research and development of algorithms in comparative modelling, ab intio modelling and structure refinement protocols. A tremendous success has been witnessed in template-based modelling protocols, whereas strategies that involve template-free modelling still lag behind, specifically for larger proteins (>150 a.a.). Various improvements have been observed in ab initio protein structure prediction methodologies overtime, with recent ones attributed to the usage of deep learning approaches to construct protein backbone structure from its amino acid sequence. This review highlights the major strategies undertaken for template-free modelling of protein structures while discussing few tools developed under each strategy. It will also briefly comment on the progress observed in the field of ab initio modelling of proteins over the course of time as seen through the evolution of CASP platform.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Nagaraja, Anamya Ajjolli; Charton, Philippe; Cadet, Xavier F; Fontaine, Nicolas; Delsaut, Mathieu; Wiltschi, Birgit; Voit, Alena; Offmann, Bernard; Damour, Cedric; Grondin-Perez, Brigitte; Cadet, Frederic
A Machine Learning Approach for Efficient Selection of Enzyme Concentrations and Its Application for Flux Optimization Article de journal
Dans: Catalysts, vol. 10, no. 3, 2020, ISSN: 2073-4344.
@article{catal10030291,
title = {A Machine Learning Approach for Efficient Selection of Enzyme Concentrations and Its Application for Flux Optimization},
author = {Anamya Ajjolli Nagaraja and Philippe Charton and Xavier F Cadet and Nicolas Fontaine and Mathieu Delsaut and Birgit Wiltschi and Alena Voit and Bernard Offmann and Cedric Damour and Brigitte Grondin-Perez and Frederic Cadet},
url = {https://www.mdpi.com/2073-4344/10/3/291},
doi = {10.3390/catal10030291},
issn = {2073-4344},
year = {2020},
date = {2020-01-01},
journal = {Catalysts},
volume = {10},
number = {3},
abstract = {The metabolic engineering of pathways has been used extensively to produce molecules of interest on an industrial scale. Methods like gene regulation or substrate channeling helped to improve the desired product yield. Cell-free systems are used to overcome the weaknesses of engineered strains. One of the challenges in a cell-free system is selecting the optimized enzyme concentration for optimal yield. Here, a machine learning approach is used to select the enzyme concentration for the upper part of glycolysis. The artificial neural network approach (ANN) is known to be inefficient in extrapolating predictions outside the box: high predicted values will bump into a sort of “glass ceiling”. In order to explore this “glass ceiling” space, we developed a new methodology named glass ceiling ANN (GC-ANN). Principal component analysis (PCA) and data classification methods are used to derive a rule for a high flux, and ANN to predict the flux through the pathway using the input data of 121 balances of four enzymes in the upper part of glycolysis. The outcomes of this study are i. in silico selection of optimum enzyme concentrations for a maximum flux through the pathway and ii. experimental in vitro validation of the “out-of-the-box” fluxes predicted using this new approach. Surprisingly, flux improvements of up to 63% were obtained. Gratifyingly, these improvements are coupled with a cost decrease of up to 25% for the assay.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Liu, Guoxia; Xuan, Ning; Rajashekar, Balaji; Arnaud, Philippe; Offmann, Bernard; Picimbon, Jean-François
Comprehensive History of CSP Genes: Evolution, Phylogenetic Distribution and Functions Article de journal
Dans: Genes, vol. 11, no. 4, p. 413, 2020.
@article{liu2020comprehensive,
title = {Comprehensive History of CSP Genes: Evolution, Phylogenetic Distribution and Functions},
author = {Guoxia Liu and Ning Xuan and Balaji Rajashekar and Philippe Arnaud and Bernard Offmann and Jean-François Picimbon},
doi = {10.3390/genes11040413},
year = {2020},
date = {2020-01-01},
journal = {Genes},
volume = {11},
number = {4},
pages = {413},
publisher = {Multidisciplinary Digital Publishing Institute},
abstract = {In this review we present the developmental, histological, evolutionary and functional properties of insect chemosensory proteins (CSPs) in insect species. CSPs are small globular proteins folded like a prism and notoriously known for their complex and arguably obscure function(s), particularly in pheromone olfaction. Here, we focus on direct functional consequences on protein function depending on duplication, expression and RNA editing. The result of our analysis is important for understanding the significance of RNA-editing on functionality of CSP genes, particularly in the brain tissue.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Téletchéa, Stéphane; Téletchéa, Fabrice
STOREFISH 2.0: a database on the reproductive strategies of teleost fishes Article de journal
Dans: Database, vol. 2020, 2020, ISSN: 1758-0463, (baaa095).
@article{10.1093/database/baaa095,
title = {STOREFISH 2.0: a database on the reproductive strategies of teleost fishes},
author = {Stéphane Téletchéa and Fabrice Téletchéa},
url = {https://doi.org/10.1093/database/baaa095},
doi = {10.1093/database/baaa095},
issn = {1758-0463},
year = {2020},
date = {2020-01-01},
journal = {Database},
volume = {2020},
abstract = {Teleost fishes show the most outstanding reproductive diversity of all vertebrates. Yet to date, no one has been able to decisively explain this striking variability nor to perform large-scale phylogenetic analyses of reproductive modes. Here, we describe STrategies Of REproduction in FISH (STOREFISH) 2.0, an online database easing the sharing of an original data set on reproduction published in 2007, enriched with automated data extraction and presentation to display the knowledge acquired on temperate freshwater fish species. STOREFISH 2.0 contains the information for 80 freshwater fish species and 50 traits from the analysis of 1219 references. It is anticipated that this new database could be useful for freshwater biodiversity research, conservation, assessment and management.Database URL: www.storefish.org},
note = {baaa095},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Dussouy, Christophe; Téletchéa, Stéphane; Lambert, Annie; Charlier, Cathy; Botez, Iuliana; Ceuninck, Frédéric De; Grandjean, Cyrille
Access to Galectin-3 Inhibitors from Chemoenzymatic Synthons Article de journal
Dans: The Journal of Organic Chemistry, vol. 85, no. 24, p. 16099-16114, 2020, (PMID: 33200927).
@article{doi:10.1021/acs.joc.0c01927b,
title = {Access to Galectin-3 Inhibitors from Chemoenzymatic Synthons},
author = {Christophe Dussouy and Stéphane Téletchéa and Annie Lambert and Cathy Charlier and Iuliana Botez and Frédéric De Ceuninck and Cyrille Grandjean},
url = {https://doi.org/10.1021/acs.joc.0c01927},
doi = {10.1021/acs.joc.0c01927},
year = {2020},
date = {2020-01-01},
journal = {The Journal of Organic Chemistry},
volume = {85},
number = {24},
pages = {16099-16114},
abstract = {Chemoenzymatic strategies are useful for providing both regio- and stereoselective access to bioactive oligosaccharides. We show herein that a glycosynthase mutant of a Thermus thermophilus α-glycosidase can react with unnatural glycosides such as 6-azido-6-deoxy-d-glucose/glucosamine to lead to β-d-galactopyranosyl-(1→3)-d-glucopyranoside or β-d-galactopyranosyl-(1→3)-2-acetamido-2-deoxy-d-glucopyranoside derivatives bearing a unique azide function. Taking advantage of the orthogonality between the azide and the hydroxyl functional groups, the former was next selectively reacted to give rise to a library of galectin-3 inhibitors. Combining enzyme substrate promiscuity and bioorthogonality thus appears as a powerful strategy to rapidly access to sugar-based ligands},
note = {PMID: 33200927},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
12 publications
Gheyouche, Ennys; Launay, Romain; Lethiec, Jean; Labeeuw, Antoine; Roze, Caroline; Amossé, Alan; Téletchéa, Stéphane
Docknmine, a web portal to assemble and analyse virtual and experimental interaction data Article de journal
Dans: International Journal of Molecular Sciences, vol. 20, no. 20, 2019, ISSN: 14220067.
@article{Gheyouche2019,
title = {Docknmine, a web portal to assemble and analyse virtual and experimental interaction data},
author = {Ennys Gheyouche and Romain Launay and Jean Lethiec and Antoine Labeeuw and Caroline Roze and Alan Amossé and Stéphane Téletchéa},
doi = {10.3390/ijms20205062},
issn = {14220067},
year = {2019},
date = {2019-10-01},
journal = {International Journal of Molecular Sciences},
volume = {20},
number = {20},
publisher = {MDPI AG},
abstract = {Scientists have to perform multiple experiments producing qualitative and quantitative data to determine if a compound is able to bind to a given target. Due to the large diversity of the potential ligand chemical space, the possibility of experimentally exploring a lot of compounds on a target rapidly becomes out of reach. Scientists therefore need to use virtual screening methods to determine the putative binding mode of ligands on a protein and then post-process the raw docking experiments with a dedicated scoring function in relation with experimental data. Two of the major difficulties for comparing docking predictions with experiments mostly come from the lack of transferability of experimental data and the lack of standardisation in molecule names. Although large portals like PubChem or ChEMBL are available for general purpose, there is no service allowing a formal expert annotation of both experimental data and docking studies. To address these issues, researchers build their own collection of data in flat files, often in spreadsheets, with limited possibilities of extensive annotations or standardisation of ligand descriptions allowing cross-database retrieval. We have conceived the dockNmine platform to provide a service allowing an expert and authenticated annotation of ligands and targets. First, this portal allows a scientist to incorporate controlled information in the database using reference identifiers for the protein (Uniprot ID) and the ligand (SMILES description), the data and the publication associated to it. Second, it allows the incorporation of docking experiments using forms that automatically parse useful parameters and results. Last, the web interface provides a lot of pre-computed outputs to assess the degree of correlations between docking experiments and experimental data.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Tripathi, Neha; Vetrivel, Iyanar; Téletchéa, Stéphane; Jean, Mickaël; Legembre, Patrick; Laurent, Adèle D
Investigation of phospholipase Cγ1 interaction with SLP76 using molecular modeling methods for identifying novel inhibitors Article de journal
Dans: International Journal of Molecular Sciences, vol. 20, no. 19, 2019, ISSN: 14220067.
@article{Tripathi2019,
title = {Investigation of phospholipase Cγ1 interaction with SLP76 using molecular modeling methods for identifying novel inhibitors},
author = {Neha Tripathi and Iyanar Vetrivel and Stéphane Téletchéa and Mickaël Jean and Patrick Legembre and Adèle D Laurent},
doi = {10.3390/ijms20194721},
issn = {14220067},
year = {2019},
date = {2019-10-01},
journal = {International Journal of Molecular Sciences},
volume = {20},
number = {19},
publisher = {MDPI AG},
abstract = {The enzyme phospholipase C gamma 1 (PLCγ1) has been identified as a potential drug target of interest for various pathological conditions such as immune disorders, systemic lupus erythematosus, and cancers. Targeting its SH3 domain has been recognized as an efficient pharmacological approach for drug discovery against PLCγ1. Therefore, for the first time, a combination of various biophysical methods has been employed to shed light on the atomistic interactions between PLCγ1 and its known binding partners. Indeed, molecular modeling of PLCγ1 with SLP76 peptide and with previously reported inhibitors (ritonavir, anethole, daunorubicin, diflunisal, and rosiglitazone) facilitated the identification of the common critical residues (Gln805, Arg806, Asp808, Glu809, Asp825, Gly827, and Trp828) as well as the quantification of their interaction through binding energies calculations. These features are in agreement with previous experimental data. Such an in depth biophysical analysis of each complex provides an opportunity to identify new inhibitors through pharmacophore mapping, molecular docking and MD simulations. From such a systematic procedure, a total of seven compounds emerged as promising inhibitors, all characterized by a strong binding with PLCγ1 and a comparable or higher binding affinity to ritonavir (ΔGbind < -25 kcal/mol), one of the most potent inhibitor reported till now.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Nagaraja, Anamya Ajjolli; Fontaine, Nicolas; Delsaut, Mathieu; Charton, Philippe; Damour, Cedric; Offmann, Bernard; Grondin-Perez, Brigitte; Cadet, Frédéric
Flux prediction using artificial neural network (ANN) for the upper part of glycolysis Article de journal
Dans: PLOS ONE, vol. 14, no. 5, p. e0216178, 2019, ISSN: 1932-6203.
@article{AjjolliNagaraja2019,
title = {Flux prediction using artificial neural network (ANN) for the upper part of glycolysis},
author = {Anamya {Ajjolli Nagaraja} and Nicolas Fontaine and Mathieu Delsaut and Philippe Charton and Cedric Damour and Bernard Offmann and Brigitte Grondin-Perez and Frédéric Cadet},
editor = {Marie-Joelle Virolle},
url = {https://dx.plos.org/10.1371/journal.pone.0216178},
doi = {10.1371/journal.pone.0216178},
issn = {1932-6203},
year = {2019},
date = {2019-05-01},
journal = {PLOS ONE},
volume = {14},
number = {5},
pages = {e0216178},
publisher = {Public Library of Science},
abstract = {The selection of optimal enzyme concentration in multienzyme cascade reactions for the highest product yield in practice is very expensive and time-consuming process. The modelling of biological pathways is a difficult process because of the complexity of the system. The mathematical modelling of the system using an analytical approach depends on the many parameters of enzymes which rely on tedious and expensive experiments. The artificial neural network (ANN) method has been successively applied in different fields of science to perform complex functions. In this study, ANN models were trained to predict the flux for the upper part of glycolysis as inferred by NADH consumption, using four enzyme concentrations i.e., phosphoglucoisomerase, phosphofructokinase, fructose-bisphosphate-aldolase, triose-phosphate-isomerase. Out of three ANN algorithms, the neuralnet package with two activation functions, “logistic” and “tanh” were implemented. The prediction of the flux was very efficient: RMSE and R2 were 0.847, 0.93 and 0.804, 0.94 respectively for logistic and tanh functions using a cross validation procedure. This study showed that a systemic approach such as ANN could be used for accurate prediction of the flux through the metabolic pathway. This could help to save a lot of time and costs, particularly from an industrial perspective. The R-code is available at: https://github.com/DSIMB/ANN-Glycolysis-Flux-Prediction.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Téletchéa, Stéphane; Santuz, H; Léonard, S; Etchebest, Catherine
Repository of enriched structures of proteins involved in the red blood cell environment (RESPIRE) Article de journal
Dans: PLoS ONE, vol. 14, no. 2, 2019, ISSN: 19326203.
@article{Teletchea2019,
title = {Repository of enriched structures of proteins involved in the red blood cell environment (RESPIRE)},
author = {Stéphane Téletchéa and H Santuz and S Léonard and Catherine Etchebest},
doi = {10.1371/journal.pone.0211043},
issn = {19326203},
year = {2019},
date = {2019-02-01},
journal = {PLoS ONE},
volume = {14},
number = {2},
publisher = {Public Library of Science},
abstract = {The Red Blood Cell (RBC) is a metabolically-driven cell vital for processes such a gas transport and homeostasis. RBC possesses at its surface exposing antigens proteins that are critical in blood transfusion. Due to their importance, numerous studies address the cell function as a whole but more and more details of RBC structure and protein content are now studied using massive state-of-the art characterisation techniques. Yet, the resulting information is frequently scattered in many scientific articles, in many databases and specialized web servers. To provide a more compendious view of erythrocytes and of their protein content, we developed a dedicated database called RESPIRE that aims at gathering a comprehensive and coherent ensemble of information and data about proteins in RBC. This cell-driven database lists proteins found in erythrocytes. For a given protein entry, initial data are processed from external portals and enriched by using state-of-the-art bioinformatics methods. As structural information is extremely useful to understand protein function and predict the impact of mutations, a strong effort has been put on the prediction of protein structures with a special treatment for membrane proteins. Browsing the database is available through text search for reference gene names or protein identifiers, through pre-defined queries or via hyperlinks. The RESPIRE database provides valuable information and unique annotations that should be useful to a wide audience of biologists, clinicians and structural biologists.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Caignec, Cédric Le; Ory, Benjamin; Lamoureux, François; O'Donohue, Marie Francoise; Orgebin, Emilien; Lindenbaum, Pierre; Téletchéa, Stéphane; Saby, Manon; Hurst, Anna; Nelson, Katherine; Gilbert, Shawn R; Wilnai, Yael; Zeitlin, Leonid; Segev, Eitan; Tesfaye, Robel; Nizon, Mathilde; Cogne, Benjamin; Bezieau, Stéphane; Geoffroy, Loic; Hamel, Antoine; Mayrargue, Emmanuelle; de Courtivron, Benoît; Decock-Giraudaud, Aliette; Charrier, Céline; Pichon, Olivier; Retière, Christelle; Redon, Richard; Pepler, Alexander; McWalter, Kirsty; Costa, Lydie Da; Toutain, Annick; Gleizes, Pierre Emmanuel; Baud'huin, Marc; Isidor, Bertrand
RPL13 Variants Cause Spondyloepimetaphyseal Dysplasia with Severe Short Stature Article de journal
Dans: American Journal of Human Genetics, vol. 105, no. 5, p. 1040–1047, 2019, ISSN: 15376605.
@article{LeCaignec2019,
title = {RPL13 Variants Cause Spondyloepimetaphyseal Dysplasia with Severe Short Stature},
author = {Cédric Le Caignec and Benjamin Ory and François Lamoureux and Marie Francoise O'Donohue and Emilien Orgebin and Pierre Lindenbaum and Stéphane Téletchéa and Manon Saby and Anna Hurst and Katherine Nelson and Shawn R Gilbert and Yael Wilnai and Leonid Zeitlin and Eitan Segev and Robel Tesfaye and Mathilde Nizon and Benjamin Cogne and Stéphane Bezieau and Loic Geoffroy and Antoine Hamel and Emmanuelle Mayrargue and Beno{î}t de Courtivron and Aliette Decock-Giraudaud and Céline Charrier and Olivier Pichon and Christelle Retière and Richard Redon and Alexander Pepler and Kirsty McWalter and Lydie Da Costa and Annick Toutain and Pierre Emmanuel Gleizes and Marc Baud'huin and Bertrand Isidor},
doi = {10.1016/j.ajhg.2019.09.024},
issn = {15376605},
year = {2019},
date = {2019-01-01},
journal = {American Journal of Human Genetics},
volume = {105},
number = {5},
pages = {1040--1047},
abstract = {Variants in genes encoding ribosomal proteins have thus far been associated with Diamond-Blackfan anemia, a rare inherited bone marrow failure, and isolated congenital asplenia. Here, we report one de novo missense variant and three de novo splice variants in RPL13, which encodes ribosomal protein RPL13 (also called eL13), in four unrelated individuals with a rare bone dysplasia causing severe short stature. The three splice variants (c.477+1GtextgreaterT, c.477+1GtextgreaterA, and c.477+2 TtextgreaterC) result in partial intron retention, which leads to an 18-amino acid insertion. In contrast to observations from Diamond-Blackfan anemia, we detected no evidence of significant pre-rRNA processing disturbance in cells derived from two affected individuals. Consistently, we showed that the insertion-containing protein is stably expressed and incorporated into 60S subunits similar to the wild-type protein. Erythroid proliferation in culture and ribosome profile on sucrose gradient are modified, suggesting a change in translation dynamics. We also provide evidence that RPL13 is present at high levels in chondrocytes and osteoblasts in mouse growth plates. Taken together, we show that the identified RPL13 variants cause a human ribosomopathy defined by a rare skeletal dysplasia, and we highlight the role of this ribosomal protein in bone development.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Ghosh, Pritha; Joshi, Adwait; Guita, Niang; Offmann, Bernard; Sowdhamini, Ramanathan
EcRBPome: A comprehensive database of all known E. coli RNA-binding proteins Article de journal
Dans: BMC Genomics, vol. 20, no. 1, p. 1–6, 2019, ISSN: 14712164.
@article{Ghosh2019,
title = {EcRBPome: A comprehensive database of all known E. coli RNA-binding proteins},
author = {Pritha Ghosh and Adwait Joshi and Niang Guita and Bernard Offmann and Ramanathan Sowdhamini},
doi = {10.1186/s12864-019-5755-5},
issn = {14712164},
year = {2019},
date = {2019-01-01},
journal = {BMC Genomics},
volume = {20},
number = {1},
pages = {1--6},
publisher = {BMC Genomics},
abstract = {The repertoire of RNA-binding proteins (RBPs) in bacteria play a crucial role in their survival, and interactions with the host machinery, but there is little information, record or characterisation in bacterial genomes. As a first step towards this, we have chosen the bacterial model system Escherichia coli, and organised all RBPs in this organism into a comprehensive database named EcRBPome. It contains RBPs recorded from 614 complete E. coli proteomes available in the RefSeq database (as of October 2018). The database provides various features related to the E. coli RBPs, like their domain architectures, PDB structures, GO and EC annotations etc. It provides the assembly, bioproject and biosample details of each strain, as well as cross-strain comparison of occurrences of various RNA-binding domains (RBDs). The percentage of RBPs, the abundance of the various RBDs harboured by each strain have been graphically represented in this database and available alongside other files for user download. To the best of our knowledge, this is the first database of its kind and we hope that it will be of great use to the biological community.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
David, Benoit; Arnaud, Philippe; Tellier, Charles; Sanejouand, Yves-Henri
Toward the design of efficient transglycosidases: the case of the GH1 of Thermus thermophilus Article de journal
Dans: Protein Engineering, Design and Selection, vol. 32, no. 7, p. 309–316, 2019, ISSN: 1741-0126.
@article{10.1093/protein/gzz032,
title = {Toward the design of efficient transglycosidases: the case of the GH1 of Thermus thermophilus},
author = {Benoit David and Philippe Arnaud and Charles Tellier and Yves-Henri Sanejouand},
url = {https://doi.org/10.1093/protein/gzz032},
doi = {10.1093/protein/gzz032},
issn = {1741-0126},
year = {2019},
date = {2019-01-01},
journal = {Protein Engineering, Design and Selection},
volume = {32},
number = {7},
pages = {309--316},
abstract = {Using the information available in the sequences of well-characterized transglycosidases found in plants, mutations were introduced in the glycoside hydrolase of the bacterium Thermus thermophilus, with the aim of turning it into an efficient transglycosidase. All mutants happen to have fair catalytic efficiencies, being at worst 25 times less efficient than the wild type. Noteworthy, W120F, one of our high transglycosylation yield (≈ 50%) mutants, is only two times less efficient than the wild type. Interestingly, while in the wild type the sidechain of the acid–base is only found able to sample a pair of equivalent conformations during 0.5-µs-long molecular dynamics simulations, its flexibility is much higher in the case of the high transglycosylation yield mutants. Our results thus suggest that engineering the flexibility of the acid–base of a retaining glycoside hydrolase could be a general way to turn it into an efficient transglycosidase.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Chaaya, Nancy; Shahsavarian, Melody A; Maffucci, Irene; Friboulet, Alain; Offmann, Bernard; Léger, Jean Benoist; Rousseau, Sylvain; Avalle, Bérangère; Padiolleau-Lefèvre, Séverine
Genetic background and immunological status influence B cell repertoire diversity in mice Article de journal
Dans: Scientific Reports, vol. 9, no. 1, p. 1–7, 2019, ISSN: 20452322.
@article{Chaaya2019,
title = {Genetic background and immunological status influence B cell repertoire diversity in mice},
author = {Nancy Chaaya and Melody A Shahsavarian and Irene Maffucci and Alain Friboulet and Bernard Offmann and Jean Benoist Léger and Sylvain Rousseau and Bérang{è}re Avalle and Séverine Padiolleau-Lef{è}vre},
doi = {10.1038/s41598-019-50714-y},
issn = {20452322},
year = {2019},
date = {2019-01-01},
journal = {Scientific Reports},
volume = {9},
number = {1},
pages = {1--7},
abstract = {The relationship between the immune repertoire and the physiopathological status of individuals is essential to apprehend the genesis and the evolution of numerous pathologies. Nevertheless, the methodological approaches to understand these complex interactions are challenging. We performed a study evaluating the diversity harbored by different immune repertoires as a function of their physiopathological status. In this study, we base our analysis on a murine scFv library previously described and representing four different immune repertoires: i) healthy and naïve, ii) healthy and immunized, iii) autoimmune prone and naïve, and iv) autoimmune prone and immunized. This library, 2.6 × 109 in size, is submitted to high throughput sequencing (Next Generation Sequencing, NGS) in order to analyze the gene subgroups encoding for immunoglobulins. A comparative study of the distribution of immunoglobulin gene subgroups present in the four libraries has revealed shifts in the B cell repertoire originating from differences in genetic background and immunological status of mice.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Vetrivel, Iyanar; de Brevern, Alexandre G; Cadet, Frédéric; Srinivasan, Narayanaswamy; Offmann, Bernard
Structural variations within proteins can be as large as variations observed across their homologues Article de journal
Dans: Biochimie, vol. 167, p. 162–170, 2019, ISSN: 61831638.
@article{Vetrivel2019,
title = {Structural variations within proteins can be as large as variations observed across their homologues},
author = {Iyanar Vetrivel and Alexandre G de Brevern and Frédéric Cadet and Narayanaswamy Srinivasan and Bernard Offmann},
doi = {10.1016/j.biochi.2019.09.013},
issn = {61831638},
year = {2019},
date = {2019-01-01},
journal = {Biochimie},
volume = {167},
pages = {162--170},
abstract = {Understanding the structural plasticity of proteins is key to understanding the intricacies of their functions and mechanistic basis. In the current study, we analyzed the available multiple crystal structures of the same protein for the structural differences. For this purpose we used an abstraction of protein structures referred as Protein Blocks (PBs) that was previously established. We also characterized the nature of the structural variations for a few proteins using molecular dynamics simulations. In both the cases, the structural variations were summarized in the form of substitution matrices of PBs. We show that certain conformational states are preferably replaced by other specific conformational states. Interestingly, these structural variations are highly similar to those previously observed across structures of homologous proteins (r2 = 0.923) or across the ensemble of conformations from NMR data (r2 = 0.919). Thus our study quantitatively shows that overall trends of structural changes in a given protein are nearly identical to the trends of structural differences that occur in the topologically equivalent positions in homologous proteins. Specific case studies are used to illustrate the nature of these structural variations.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Richoux, Florian; Servantie, Charlène; Borès, Cynthia; Téletchéa, Stéphane
Comparing two deep learning sequence-based models for protein-protein interaction prediction Article de journal
Dans: arXiv, vol. 1901.06268, 2019.
@article{Richoux2019,
title = {Comparing two deep learning sequence-based models for protein-protein interaction prediction},
author = {Florian Richoux and Charlène Servantie and Cynthia Borès and Stéphane Téletchéa},
url = {http://arxiv.org/abs/1901.06268},
year = {2019},
date = {2019-01-01},
journal = {arXiv},
volume = {1901.06268},
abstract = {Biological data are extremely diverse, complex but also quite sparse. The recent developments in deep learning methods are offering new possibilities for the analysis of complex data. However, it is easy to be get a deep learning model that seems to have good results but is in fact either overfitting the training data or the validation data. In particular, the fact to overfit the validation data, called "information leak", is almost never treated in papers proposing deep learning models to predict protein-protein interactions (PPI). In this work, we compare two carefully designed deep learning models and show pitfalls to avoid while predicting PPIs through machine learning methods. Our best model predicts accurately more than 78% of human PPI, in very strict conditions both for training and testing. The methodology we propose here allow us to have strong confidences about the ability of a model to scale up on larger datasets. This would allow sharper models when larger datasets would be available, rather than current models prone to information leaks. Our solid methodological foundations shall be applicable to more organisms and whole proteome networks predictions.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Vetrivel, Iyanar; Hoffmann, Lionel; Guegan, Sean; Offmann, Bernard; Laurent, Adele D
PBmapclust: Mapping and Clustering the Protein Conformational Space Using a Structural Alphabet Proceedings Article
Dans: Byska, Jan; Krone, Michael; Sommer, Björn (Ed.): Workshop on Molecular Graphics and Visual Analysis of Molecular Data, The Eurographics Association, 2019, ISBN: 978-3-03868-085-7.
@inproceedings{lva.20191097b,
title = {PBmapclust: Mapping and Clustering the Protein Conformational Space Using a Structural Alphabet},
author = {Iyanar Vetrivel and Lionel Hoffmann and Sean Guegan and Bernard Offmann and Adele D Laurent},
editor = {Jan Byska and Michael Krone and Björn Sommer},
doi = {10.2312/molva.20191097},
isbn = {978-3-03868-085-7},
year = {2019},
date = {2019-01-01},
booktitle = {Workshop on Molecular Graphics and Visual Analysis of Molecular Data},
publisher = {The Eurographics Association},
abstract = {Analyzing the data from molecular dynamics simulation of biological macromolecules like proteins is challenging. We propose a simple tool called PBmapclust that is based on a well established structural alphabet called Protein blocks (PB). PBs help in tracing the trajectory of the protein backbone by categorizing it into 16 distinct structural states. PBmapclust provides a time vs. amino acid residue plot that is color coded to match each of the PBs. Color changes correspond to structural changes, giving a visual overview of the simulation. Further, PBmapclust enables the user to "map" the conformational space sampled by the protein during the MD simulation by clustering the conformations. The ability to generate sub-maps for specific residues and specific time intervals allows the user to focus on residues of interest like for active sites or disordered regions. We have included an illustrative case study to demonstrate the utility of the tool. It describes the effect of the disordered domain of a HSP90 co-chaperone on the conformation of its active site residues. The scripts required to perform PBmapclust are made freely available under the GNU general public license.},
keywords = {},
pubstate = {published},
tppubtype = {inproceedings}
}
Liu, Guoxia; Arnaud, Philippe; Offmann, Bernard; Picimbon, Jean-François
Pheromone, Natural Odor and Odorant Reception Suppressing Agent (ORSA) for Insect Control Book Section
Dans: Olfactory Concepts of Insect Control-Alternative to Insecticides, p. 311–345, Springer, Cham, 2019.
@incollection{liu2019pheromone,
title = {Pheromone, Natural Odor and Odorant Reception Suppressing Agent (ORSA) for Insect Control},
author = {Guoxia Liu and Philippe Arnaud and Bernard Offmann and Jean-François Picimbon},
doi = {10.1007/978-3-030-05165-5},
year = {2019},
date = {2019-01-01},
booktitle = {Olfactory Concepts of Insect Control-Alternative to Insecticides},
pages = {311--345},
publisher = {Springer, Cham},
abstract = {Odorant-binding proteins (OBPs) are small ``bowl-like'' globular pro- teins, highly abundant in the antennae of most insect species. These proteins are believed to mediate reception of odor molecules at the periphery of sensory receptor neurons. Therefore, they may represent crucial targets for becoming new methods of insect pest control by directly interfering with the olfactory acuity of the insect. The current better understanding of molecular mechanisms underlying odor detec- tion and the knowledge about the functional binding sites of OBPs and many other families of binding proteins in various insect species is elucidated here. Such infor- mation forms the basis for the synthesis of new inhibitor olfactory compounds (Odorant Reception-Suppressing Agents, ORSAs) to interact specifically with the groups of insect pests.},
keywords = {},
pubstate = {published},
tppubtype = {incollection}
}
1 publication
Cadet, Frédéric; Fontaine, Nicolas; Li, Guangyue; Sanchis, Joaquin; Chong, Matthieu Ng Fuk; Pandjaitan, Rudy; Vetrivel, Iyanar; Offmann, Bernard; Reetz, Manfred T
A machine learning approach for reliable prediction of amino acid interactions and its application in the directed evolution of enantioselective enzymes Article de journal
Dans: Scientific Reports, vol. 8, no. 1, p. 1–15, 2018, ISSN: 20452322.
@article{Cadet2018,
title = {A machine learning approach for reliable prediction of amino acid interactions and its application in the directed evolution of enantioselective enzymes},
author = {Frédéric Cadet and Nicolas Fontaine and Guangyue Li and Joaquin Sanchis and Matthieu {Ng Fuk Chong} and Rudy Pandjaitan and Iyanar Vetrivel and Bernard Offmann and Manfred T Reetz},
doi = {10.1038/s41598-018-35033-y},
issn = {20452322},
year = {2018},
date = {2018-01-01},
journal = {Scientific Reports},
volume = {8},
number = {1},
pages = {1--15},
abstract = {Directed evolution is an important research activity in synthetic biology and biotechnology. Numerous reports describe the application of tedious mutation/screening cycles for the improvement of proteins. Recently, knowledge-based approaches have facilitated the prediction of protein properties and the identification of improved mutants. However, epistatic phenomena constitute an obstacle which can impair the predictions in protein engineering. We present an innovative sequence-activity relationship (innov'SAR) methodology based on digital signal processing combining wet-lab experimentation and computational protein design. In our machine learning approach, a predictive model is developed to find the resulting property of the protein when the n single point mutations are permuted (2n combinations). The originality of our approach is that only sequence information and the fitness of mutants measured in the wet-lab are needed to build models. We illustrate the application of the approach in the case of improving the enantioselectivity of an epoxide hydrolase from Aspergillus niger. n = 9 single point mutants of the enzyme were experimentally assessed for their enantioselectivity and used as a learning dataset to build a model. Based on combinations of the 9 single point mutations (29), the enantioselectivity of these 512 variants were predicted, and candidates were experimentally checked: better mutants with higher enantioselectivity were indeed found.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
10 publications
Vetrivel, Iyanar; Mahajan, Swapnil; Tyagi, Manoj; Hoffmann, Lionel; Sanejouand, Yves-Henri; Srinivasan, Narayanaswamy; Brevern, Alexandre G De; Cadet, Frédéric; Offmann, Bernard
Knowledge-based prediction of protein backbone conformation using a structural alphabet Article de journal
Dans: PLoS ONE, vol. 12, no. 11, 2017, ISSN: 19326203.
@article{Vetrivel2017,
title = {Knowledge-based prediction of protein backbone conformation using a structural alphabet},
author = {Iyanar Vetrivel and Swapnil Mahajan and Manoj Tyagi and Lionel Hoffmann and Yves-Henri Sanejouand and Narayanaswamy Srinivasan and Alexandre G {De Brevern} and Frédéric Cadet and Bernard Offmann},
doi = {10.1371/journal.pone.0186215},
issn = {19326203},
year = {2017},
date = {2017-11-01},
journal = {PLoS ONE},
volume = {12},
number = {11},
publisher = {Public Library of Science},
abstract = {Libraries of structural prototypes that abstract protein local structures are known as structural alphabets and have proven to be very useful in various aspects of protein structure analyses and predictions. One such library, Protein Blocks, is composed of 16 standard 5-residues long structural prototypes. This form of analyzing proteins involves drafting its structure as a string of Protein Blocks. Predicting the local structure of a protein in terms of protein blocks is the general objective of this work. A new approach, PB-kPRED is proposed towards this aim. It involves (i) organizing the structural knowledge in the form of a database of pentapeptide fragments extracted from all protein structures in the PDB and (ii) applying a knowledge-based algorithm that does not rely on any secondary structure predictions and/ or sequence alignment profiles, to scan this database and predict most probable backbone conformations for the protein local structures. Though PB-kPRED uses the structural information from homologues in preference, if available. The predictions were evaluated rigorously on 15,544 query proteins representing a non-redundant subset of the PDB filtered at 30% sequence identity cut-off. We have shown that the kPRED method was able to achieve mean accuracies ranging from 40.8% to 66.3% depending on the availability of homologues. The impact of the different strategies for scanning the database on the prediction was evaluated and is discussed. Our results highlight the usefulness of the method in the context of proteins without any known structural homologues. A scoring function that gives a good estimate of the accuracy of prediction was further developed. This score estimates very well the accuracy of the algorithm (R2 of 0.82). An online version of the tool is provided freely for non-commercial usage at http://www.bo-protscience.fr/kpred/.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Sanejouand, Yves-Henri
Mutational dynamics of influenza A viruses: a principal component analysis of hemagglutinin sequences of subtype H1 Article de journal
Dans: 2017.
@article{Sanejouand2017b,
title = {Mutational dynamics of influenza A viruses: a principal component analysis of hemagglutinin sequences of subtype H1},
author = {Yves-Henri Sanejouand},
url = {http://arxiv.org/abs/1710.01594},
year = {2017},
date = {2017-10-01},
abstract = {A principal component analysis of a multiple sequence alignement of hemagglutinin sequences of subtype H1 has been performed, the sequences being encoded using the amino-acid property that maximizes the weight of the major component. In the case of this alignment, it happens to be a well-known hydrophobicity scale. Interestingly, sequences coming from human have large positive amplitudes along the major component before 2009, and large negative ones afterwards. This means that the 2009 pandemic was associated to a major change in the hydrophobicity pattern of hemagglutinin. The present analysis also highlights the high variability of viral sequences coming from swine. At a more general level, the method proposed in this paper allows to describe a sequence coming from an alignment with a set of numbers, the original point being that the choice of the corresponding property is driven by the data. This approach should allow the application of numerous methods to the study of large multiple sequence alignments.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Sanejouand, Yves-Henri
A singular mutation in the hemagglutinin of the 1918 pandemic virus Article de journal
Dans: Archives of Biochemistry and Biophysics, vol. 625-626, p. 13–16, 2017, ISSN: 0003-9861.
@article{SANEJOUAND201713,
title = {A singular mutation in the hemagglutinin of the 1918 pandemic virus},
author = {Yves-Henri Sanejouand},
url = {http://www.sciencedirect.com/science/article/pii/S0003986117302576},
doi = {https://doi.org/10.1016/j.abb.2017.05.013},
issn = {0003-9861},
year = {2017},
date = {2017-01-01},
journal = {Archives of Biochemistry and Biophysics},
volume = {625-626},
pages = {13--16},
abstract = {The influenza pandemic of 1918–1919 killed at least 50 million people. The reasons why this pandemic was so deadly remain largely unknown [9]. However, It has been shown that the 1918 viral hemagglutinin allows to reproduce the hallmarks of the illness observed during the original pandemic [11]. Thanks to the wealth of hemagglutinin sequences accumulated over the last decades, amino-acid substitutions that are found in the 1918–1919 sequences but rare otherwise can be identified with high confidence. Noteworthy, Gly 188, which is located within a key motif of the receptor binding site, has never been observed again in sequences of human viruses of subtype H1. Monitoring this singular mutation in viral sequences may help prevent another dramatic pandemic.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Mahajan, Swapnil; Sanejouand, Yves-Henri
Jumping between protein conformers using normal modes Article de journal
Dans: Journal of Computational Chemistry, vol. 38, no. 18, p. 1622–1630, 2017, ISSN: 1096987X.
@article{Mahajan2017,
title = {Jumping between protein conformers using normal modes},
author = {Swapnil Mahajan and Yves-Henri Sanejouand},
doi = {10.1002/jcc.24803},
issn = {1096987X},
year = {2017},
date = {2017-01-01},
journal = {Journal of Computational Chemistry},
volume = {38},
number = {18},
pages = {1622--1630},
abstract = {The relationship between the normal modes of a protein and its functional conformational change has been studied for decades. However, using this relationship in a predictive context remains a challenge. In this work, we demonstrate that, starting from a given protein conformer, it is possible to generate in a single step model conformers that are less than 1 Å (Cα-RMSD) from the conformer which is the known endpoint of the conformational change, particularly when the conformational change is collective in nature. Such accurate model conformers can be generated by following either the so-called robust or the 50 lowest-frequency modes obtained with various Elastic Network Models (ENMs). Interestingly, the quality of many of these models compares well with actual crystal structures, as assessed by the ROSETTA scoring function and PROCHECK. The most accurate and best quality conformers obtained in the present study were generated by using the 50 lowest-frequency modes of an all-atom ENM. However, with less than ten robust modes, which are identified without any prior knowledge of the nature of the conformational change, nearly 90% of the motion described by the 50 lowest-frequency modes of a protein can be captured. Such results strongly suggest that exploring the robust modes of ENMs may prove efficient for sampling the functionally relevant conformational repertoire of many proteins. textcopyright 2017 Wiley Periodicals, Inc.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Atmanene, Cédric; Ronin, Céline; Téletchéa, Stéphane; Gautier, François Moana; Djedaïni-Pilard, Florence; Ciesielski, Fabrice; Vivat, Valérie; Grandjean, Cyrille
Biophysical and structural characterization of mono/di-arylated lactosamine derivatives interaction with human galectin-3 Article de journal
Dans: Biochemical and Biophysical Research Communications, vol. 489, no. 3, p. 281–286, 2017, ISSN: 10902104.
@article{Atmanene2017,
title = {Biophysical and structural characterization of mono/di-arylated lactosamine derivatives interaction with human galectin-3},
author = {Cédric Atmanene and Céline Ronin and Stéphane Téletchéa and François Moana Gautier and Florence Djedaïni-Pilard and Fabrice Ciesielski and Valérie Vivat and Cyrille Grandjean},
doi = {10.1016/j.bbrc.2017.05.150},
issn = {10902104},
year = {2017},
date = {2017-01-01},
journal = {Biochemical and Biophysical Research Communications},
volume = {489},
number = {3},
pages = {281--286},
abstract = {Combination of biophysical and structural techniques allowed characterizing and uncovering the mechanisms underlying increased binding affinity of lactosamine derivatives for galectin 3. In particular, complementing information gathered from X-ray crystallography, native mass spectrometry and isothermal microcalorimetry showed favorable enthalpic contribution of cation-π interaction between lactosamine aryl substitutions and arginine residues from the carbohydrate recognition domain, which resulted in two log increase in compound binding affinity. This incrementing strategy allowed individual contribution of galectin inhibitor moieties to be dissected. Altogether, our results suggest that core and substituents of these saccharide-based inhibitors can be optimized separately, providing valuable tools to study the role of galectins in diseases.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Labbé, Pauline; Faure, Emilie; Lecointe, Simon; Scouarnec, Solena Le; Kyndt, Florence; Marrec, Marie; Tourneau, Thierry Le; Offmann, Bernard; Duplaà, Cécile; Zaffran, Stéphane; Schott, Jean Jacques; Merot, Jean
The alternatively spliced LRRFIP1 Isoform-1 is a key regulator of the Wnt/β-catenin transcription pathway Article de journal
Dans: Biochimica et Biophysica Acta - Molecular Cell Research, vol. 1864, no. 7, p. 1142–1152, 2017, ISSN: 18792596.
@article{Labbe2017,
title = {The alternatively spliced LRRFIP1 Isoform-1 is a key regulator of the Wnt/β-catenin transcription pathway},
author = {Pauline Labbé and Emilie Faure and Simon Lecointe and Solena {Le Scouarnec} and Florence Kyndt and Marie Marrec and Thierry {Le Tourneau} and Bernard Offmann and Cécile Duplaà and Stéphane Zaffran and Jean Jacques Schott and Jean Merot},
doi = {10.1016/j.bbamcr.2017.03.008},
issn = {18792596},
year = {2017},
date = {2017-01-01},
journal = {Biochimica et Biophysica Acta - Molecular Cell Research},
volume = {1864},
number = {7},
pages = {1142--1152},
abstract = {The GC-rich Binding Factor 2/Leucine Rich Repeat in the Flightless 1 Interaction Protein 1 gene (GCF2/LRRFIP1) is predicted to be alternatively spliced in five different isoforms. Although important peptide sequence differences are expected to result from this alternative splicing, to date, only the gene transcription regulator properties of LRRFIP1-Iso5 were unveiled. Based on molecular, cellular and biochemical data, we show here that the five isoforms define two molecular entities with different expression profiles in human tissues, subcellular localizations, oligomerization properties and transcription enhancer properties of the canonical Wnt pathway. We demonstrated that LRRFIP1-Iso3, -4 and -5, which share over 80% sequence identity, are primarily located in the cell cytoplasm and form homo and hetero-multimers between each other. In contrast, LRRFIP1-Iso1 and -2 are primarily located in the cell nucleus in part thanks to their shared C-terminal domain. Furthermore, we showed that LRRFIP1-Iso1 is preferentially expressed in the myocardium and skeletal muscle. Using the in vitro Topflash reporter assay we revealed that among LRRFIP1 isoforms, LRRFIP1-Iso1 is the strongest enhancer of the β-catenin Wnt canonical transcription pathway thanks to a specific N-terminal domain harboring two critical tryptophan residues (W76, 82). In addition, we showed that the Wnt enhancer properties of LRRFIP1-Iso1 depend on its homo-dimerisation which is governed by its specific coiled coil domain. Together our study identified LRRFIP1-Iso1 as a critical regulator of the Wnt canonical pathway with a potential role in myocyte differentiation and myogenesis.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
David, Benoit; Irague, Romain; Jouanneau, Diane; Daligault, Franck; Czjzek, Mirjam; Sanejouand, Yves-Henri; Tellier, Charles
Internal Water Dynamics Control the Transglycosylation/Hydrolysis Balance in the Agarase (AgaD) of Zobellia galactanivorans Article de journal
Dans: ACS Catalysis, vol. 7, no. 5, p. 3357–3367, 2017, ISSN: 21555435.
@article{David2017a,
title = {Internal Water Dynamics Control the Transglycosylation/Hydrolysis Balance in the Agarase (AgaD) of Zobellia galactanivorans},
author = {Benoit David and Romain Irague and Diane Jouanneau and Franck Daligault and Mirjam Czjzek and Yves-Henri Sanejouand and Charles Tellier},
doi = {10.1021/acscatal.7b00348},
issn = {21555435},
year = {2017},
date = {2017-01-01},
journal = {ACS Catalysis},
volume = {7},
number = {5},
pages = {3357--3367},
abstract = {In retaining glycoside hydrolases (GHs), transglycosylase activity is often low due to the natural hydrolytic activity that is favored in water. Improving the relative transglycosylase activity of these enzymes is of particular interest to obtain enzymes suitable for the synthesis of oligosaccharides. We explored the effect of engineering the water dynamics within the endo-β-agarase AgaD on the transglycosylation/hydrolysis (T/H) balance. By mutating three amino acids (D341, Q342, and S351), which could control water access to a putative water channel ending close to the active site, we obtained AgaD variants with an inverted T/H balance. For the best mutant, D341L/Q342H/S351F, the hydrolysis activity was reduced 50-fold in comparison to the wild type, while the transglycosylase activity was maintained and even slightly improved. This variant produced a large amount of oligo-agaroses by a disproportionation reaction with deca-agarose as the substrate. Molecular dynamics simulations showed that these enzymatic modifications were correlated with higher water dynamics, as revealed by a marked reduction in the water survival time and a decrease in the purge time of water in a channel ending close to the active site. These results suggest that modifying the water dynamics in GHs could be a rational basis for engineering of transglycosylase activity.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Shahsavarian, Melody A; Chaaya, Nancy; Costa, Narciso; Boquet, Didier; Atkinson, Alexandre; Offmann, Bernard; Kaveri, Srini V; Lacroix-Desmazes, Sébastien; Friboulet, Alain; Avalle, Bérangère; Padiolleau-Lefèvre, Séverine
Multitarget selection of catalytic antibodies with β-lactamase activity using phage display Article de journal
Dans: FEBS Journal, vol. 284, no. 4, p. 634–653, 2017, ISSN: 17424658.
@article{Shahsavarian2017,
title = {Multitarget selection of catalytic antibodies with β-lactamase activity using phage display},
author = {Melody A Shahsavarian and Nancy Chaaya and Narciso Costa and Didier Boquet and Alexandre Atkinson and Bernard Offmann and Srini V Kaveri and Sébastien Lacroix-Desmazes and Alain Friboulet and Bérangère Avalle and Séverine Padiolleau-Lefèvre},
doi = {10.1111/febs.14012},
issn = {17424658},
year = {2017},
date = {2017-01-01},
journal = {FEBS Journal},
volume = {284},
number = {4},
pages = {634--653},
abstract = {β-lactamase enzymes responsible for bacterial resistance to antibiotics are among the most important health threats to the human population today. Understanding the increasingly vast structural motifs responsible for the catalytic mechanism of β-lactamases will help improve the future design of new generation antibiotics and mechanism-based inhibitors of these enzymes. Here we report the construction of a large murine single chain fragment variable (scFv) phage display library of size 2.7 × 109 with extended diversity by combining different mouse models. We have used two molecularly different inhibitors of the R-TEM β-lactamase as targets for selection of catalytic antibodies with β-lactamase activity. This novel methodology has led to the isolation of five antibody fragments, which are all capable of hydrolyzing the β-lactam ring. Structural modeling of the selected scFv has revealed the presence of different motifs in each of the antibody fragments potentially responsible for their catalytic activity. Our results confirm (a) the validity of using our two target inhibitors for the in vitro selection of catalytic antibodies endowed with β-lactamase activity, and (b) the plasticity of the β-lactamase active site responsible for the wide resistance of these enzymes to clinically available inhibitors and antibiotics.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Dion, Johann; Advedissian, Tamara; Storozhylova, Nataliya; Dahbi, Samir; Lambert, Annie; Deshayes, Frédérique; Viguier, Mireille; Tellier, Charles; Poirier, Françoise; Téletchéa, Stéphane; Dussouy, Christophe; Tateno, Hiroaki; Hirabayashi, Jun; Grandjean, Cyrille
Development of a Sensitive Microarray Platform for the Ranking of Galectin Inhibitors: Identification of a Selective Galectin-3 Inhibitor Article de journal
Dans: ChemBioChem, vol. 18, no. 24, p. 2428–2440, 2017, ISSN: 14397633.
@article{Dion2017a,
title = {Development of a Sensitive Microarray Platform for the Ranking of Galectin Inhibitors: Identification of a Selective Galectin-3 Inhibitor},
author = {Johann Dion and Tamara Advedissian and Nataliya Storozhylova and Samir Dahbi and Annie Lambert and Frédérique Deshayes and Mireille Viguier and Charles Tellier and Françoise Poirier and Stéphane Téletchéa and Christophe Dussouy and Hiroaki Tateno and Jun Hirabayashi and Cyrille Grandjean},
doi = {10.1002/cbic.201700544},
issn = {14397633},
year = {2017},
date = {2017-01-01},
journal = {ChemBioChem},
volume = {18},
number = {24},
pages = {2428--2440},
abstract = {Glycan microarrays are useful tools for lectin glycan profiling. The use of a glycan microarray based on evanescent-field fluorescence detection was herein further extended to the screening of lectin inhibitors in competitive experiments. The efficacy of this approach was tested with 2/3′-mono- and 2,3′-diaromatic type II lactosamine derivatives and galectins as targets and was validated by comparison with fluorescence anisotropy proposed as an orthogonal protein interaction measurement technique. We showed that subtle differences in the architecture of the inhibitor could be sensed that pointed out the preference of galectin-3 for 2′-arylamido derivatives over ureas, thioureas, and amines and that of galectin-7 for derivatives bearing an α substituent at the anomeric position of glucosamine. We eventually identified a diaromatic oxazoline as a highly specific inhibitor of galectin-3 versus galectin-1 and galectin-7.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
David, Benoît
Conception rationnelle dénzyme: conversion de glycoside hydrolases en transglycosidases Thèse
Université de Nantes, 2017.
@phdthesis{david2017conception,
title = {Conception rationnelle dénzyme: conversion de glycoside hydrolases en transglycosidases},
author = {Benoît David},
url = {https://www.theses.fr/2017NANT1044},
year = {2017},
date = {2017-01-01},
school = {Université de Nantes},
abstract = {Catalyseurs de la dégradation de polysaccharides dans le cadre de diverses applications industrielles, de nombreuses glycoside hydrolases (GH) possèdent également une activité de transglycosylation qui peut être exploitée pour la synthèse dóligosaccharides. Afin dáugmenter cette activité, minoritaire par rapport à l'hydrolyse, des expériences de mutagenèse rationnelle peuvent être employées. Toutefois, lénsemble des bases moléculaires régissant l’équilibre entre ces deux activités reste en revanche difficile a élucider. L'étude de quatre GH (Ttβgly, AgaD, TcTS, TrSA) par simulation de dynamique moléculaire a permis la découverte de canaux déau internes à leurs structures et connectant le site actif au milieu. Cette observation suggère que les canaux déau internes aux GH pourraient être impliqués dans leur activité d'hydrolyse. Plusieurs paires de résidus bordant deux de ces canaux ont été mis en évidence chez Ttβgly et AgaD et semblent contrôler le passage de léau du canal vers le site actif. La mutagenèse de ces résidus a été entreprise afin de tenter dáugmenter láctivité de transglycosylation chez ces deux enzymes. Une réduction de l'hydrolyse d'un facteur 7 et 50 au profit de láctivité de transglycosylation a été caractérisée chez les deux meilleurs mutants de Ttβgly et AgaD, respectivement. Lánalyse des simulations a révélé que ces résultats étaient corrélés à une augmentation de la dynamique des molécules déau internes aux deux canaux étudiés. Cette étude souligne ainsi límportance fonctionnelle de léau interne aux hydrolases et suggère que língénierie de sa dynamique peut constituer une approche originale pour convertir les GH en transglycosidases.},
keywords = {},
pubstate = {published},
tppubtype = {phdthesis}
}
2 publications
Liu, Guoxia; Ma, Hongmei; Xie, Hongyan; Xuan, Ning; Guo, Xia; Fan, Zhongxue; Rajashekar, Balaji; Arnaud, Philippe; Offmann, Bernard; Picimbon, Jean François
Biotype characterization, developmental profiling, insecticide response and binding property of Bemisia tabaci chemosensory proteins: Role of CSP in insect defense Article de journal
Dans: PLoS ONE, vol. 11, no. 5, 2016, ISSN: 19326203.
@article{Liu2016,
title = {Biotype characterization, developmental profiling, insecticide response and binding property of Bemisia tabaci chemosensory proteins: Role of CSP in insect defense},
author = {Guoxia Liu and Hongmei Ma and Hongyan Xie and Ning Xuan and Xia Guo and Zhongxue Fan and Balaji Rajashekar and Philippe Arnaud and Bernard Offmann and Jean François Picimbon},
doi = {10.1371/journal.pone.0154706},
issn = {19326203},
year = {2016},
date = {2016-05-01},
journal = {PLoS ONE},
volume = {11},
number = {5},
publisher = {Public Library of Science},
abstract = {Chemosensory proteins (CSPs) are believed to play a key role in the chemosensory process in insects. Sequencing genomic DNA and RNA encoding CSP1, CSP2 and CSP3 in the sweet potato whitefly Bemisia tabaci showed strong variation between B and Q biotypes. Analyzing CSP-RNA levels showed not only biotype, but also age and developmental stage-specific expression. Interestingly, applying neonicotinoid thiamethoxam insecticide using twenty-five different dose/time treatments in B and Q young adults showed that Bemisia CSP1, CSP2 and CSP3 were also differentially regulated over insecticide exposure. In our study one of the adult-specific gene (CSP1) was shown to be significantly up-regulated by the insecticide in Q, the most highly resistant form of B. tabaci. Correlatively, competitive binding assays using tryptophan fluorescence spectroscopy and molecular docking demonstrated that CSP1 protein preferentially bound to linoleic acid, while CSP2 and CSP3 proteins rather associated to another completely different type of chemical, i.e. α-pentyl-cinnamaldehyde (jasminaldehyde). This might indicate that some CSPs in whiteflies are crucial to facilitate the transport of fatty acids thus regulating some metabolic pathways of the insect immune response, while some others are tuned to much more volatile chemicals known not only for their pleasant odor scent, but also for their potent toxic insecticide activity.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Verhaeghe, Tom; Winter, Karel De; Berland, Magali; Vreese, Rob De; D'Hooghe, Matthias; Offmann, Bernard; Desmet, Tom
Converting bulk sugars into prebiotics: Semi-rational design of a transglucosylase with controlled selectivity Article de journal
Dans: Chemical Communications, vol. 52, no. 18, p. 3687–3689, 2016, ISSN: 1364548X.
@article{Verhaeghe2016,
title = {Converting bulk sugars into prebiotics: Semi-rational design of a transglucosylase with controlled selectivity},
author = {Tom Verhaeghe and Karel {De Winter} and Magali Berland and Rob {De Vreese} and Matthias D'Hooghe and Bernard Offmann and Tom Desmet},
doi = {10.1039/c5cc09940d},
issn = {1364548X},
year = {2016},
date = {2016-01-01},
journal = {Chemical Communications},
volume = {52},
number = {18},
pages = {3687--3689},
publisher = {Royal Society of Chemistry},
abstract = {Despite the growing importance of prebiotics in nutrition and gastroenterology, their structural variety is currently still very limited. The lack of straightforward procedures to gain new products in sufficient amounts often hampers application testing and further development. Although the enzyme sucrose phosphorylase can be used to produce the rare disaccharide kojibiose (α-1,2-glucobiose) from the bulk sugars sucrose and glucose, the target compound is only a side product that is difficult to isolate. Accordingly, for this biocatalyst to become economically attractive, the formation of other glucobioses should be avoided and therefore we applied semi-rational mutagenesis and low-throughput screening, which resulted in a double mutant (L341I-Q345S) with a selectivity of 95% for kojibiose. That way, an efficient and scalable production process with a yield of 74% could be established, and with a simple yeast treatment and crystallization step over a hundred grams of highly pure kojibiose (textgreater99.5%) was obtained.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
8 publications
Mahajan, Swapnil; Brevern, Alexandre G De; Sanejouand, Yves-Henri; Srinivasan, Narayanaswamy; Offmann, Bernard
Use of a structural alphabet to find compatible folds for amino acid sequences Article de journal
Dans: Protein Science, vol. 24, no. 1, p. 145–153, 2015, ISSN: 1469896X.
@article{Mahajan2015a,
title = {Use of a structural alphabet to find compatible folds for amino acid sequences},
author = {Swapnil Mahajan and Alexandre G {De Brevern} and Yves-Henri Sanejouand and Narayanaswamy Srinivasan and Bernard Offmann},
doi = {10.1002/pro.2581},
issn = {1469896X},
year = {2015},
date = {2015-01-01},
journal = {Protein Science},
volume = {24},
number = {1},
pages = {145--153},
abstract = {The structural annotation of proteins with no detectable homologs of known 3D structure identified using sequence-search methods is a major challenge today. We propose an original method that computes the conditional probabilities for the amino-acid sequence of a protein to fit to known protein 3D structures using a structural alphabet, known as "Protein Blocks" (PBs). PBs constitute a library of 16 local structural prototypes that approximate every part of protein backbone structures. It is used to encode 3D protein structures into 1D PB sequences and to capture sequence to structure relationships. Our method relies on amino acid occurrence matrices, one for each PB, to score global and local threading of query amino acid sequences to protein folds encoded into PB sequences. It does not use any information from residue contacts or sequence-search methods or explicit incorporation of hydrophobic effect. The performance of the method was assessed with independent test datasets derived from SCOP 1.75A. With a Z-score cutoff that achieved 95% specificity (i.e., less than 5% false positives), global and local threading showed sensitivity of 64.1% and 34.2%, respectively. We further tested its performance on 57 difficult CASP10 targets that had no known homologs in PDB: 38 compatible templates were identified by our approach and 66% of these hits yielded correctly predicted structures. This method scales-up well and offers promising perspectives for structural annotations at genomic level. It has been implemented in the form of a web-server that is freely available at http://www.bo-protscience.fr/forsa.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Mahajan, Swapnil; Sanejouand, Yves-Henri
On the relationship between low-frequency normal modes and the large-scale conformational changes of proteins Article de journal
Dans: Archives of Biochemistry and Biophysics, vol. 567, p. 59–65, 2015, ISSN: 10960384.
@article{Mahajan2015b,
title = {On the relationship between low-frequency normal modes and the large-scale conformational changes of proteins},
author = {Swapnil Mahajan and Yves-Henri Sanejouand},
url = {http://dx.doi.org/10.1016/j.abb.2014.12.020},
doi = {10.1016/j.abb.2014.12.020},
issn = {10960384},
year = {2015},
date = {2015-01-01},
journal = {Archives of Biochemistry and Biophysics},
volume = {567},
pages = {59--65},
publisher = {Elsevier Inc.},
abstract = {Normal mode analysis is a computational technique that allows to study the dynamics of biological macromolecules. It was first applied to small protein cases, more than thirty years ago. The interest in this technique then raised when it was realized that it can provide insights about the large-scale conformational changes a protein can experience, for instance upon ligand binding. As it was also realized that studying highly simplified protein models can provide similar insights, meaning that this kind of analysis can be both quick and simple to handle, several applications were proposed, in the context of various structural biology techniques. This review focuses on these applications, as well as on how the functional relevance of the lowest-frequency modes of proteins was established.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Teze, David; Daligault, Franck; Ferrières, Vincent; Sanejouand, Yves-Henri; Tellier, Charles
Semi-rational approach for converting a GH36 α-glycosidase into an α-transglycosidase Article de journal
Dans: Glycobiology, vol. 25, no. 4, p. 420–427, 2015, ISSN: 14602423.
@article{Teze2015,
title = {Semi-rational approach for converting a GH36 α-glycosidase into an α-transglycosidase},
author = {David Teze and Franck Daligault and Vincent Ferrières and Yves-Henri Sanejouand and Charles Tellier},
doi = {10.1093/glycob/cwu124},
issn = {14602423},
year = {2015},
date = {2015-01-01},
journal = {Glycobiology},
volume = {25},
number = {4},
pages = {420--427},
abstract = {A large number of retaining glycosidases catalyze both hydrolysis and transglycosylation reactions. In order to use them as catalysts for oligosaccharide synthesis, the balance between these two competing reactions has to be shifted toward transglycosylation. We previously designed a semi-rational approach to convert the Thermus thermophilus β-glycosidases into transglycosidases by mutating highly conserved residues located around the -1 subsite. In an attempt to verify that this strategy could be a generic approach to turn glycosidases into transglycosidases, Geobacillus stearothermophilus α-galactosidase (AgaB) was selected in order to obtain α-transgalactosidases. This is of particular interest as, to date, there are no efficient α-galactosynthases, despite the considerable importance of α-galactooligosaccharides. Thus, by site-directed mutagenesis on 14 AgaB residues, 26 single mutants and 22 double mutants were created and screened, of which 11 single mutants and 6 double mutants exhibited improved synthetic activity, producing 4-nitrophenyl α-d-galactopyranosyl-(1,6)-α-d-galactopyranoside in 26-57% yields against only 22% when native AgaB was used. It is interesting to note that the best variant was obtained by mutating a second-shell residue, with no direct interaction with the substrate or a catalytic amino acid. As this approach has proved to be efficient with both α- and β-glycosidases, it is a promising route to convert retaining glycosidases into transglycosidases.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Saumonneau, Amélie; Champion, Elise; Peltier-Pain, Pauline; Molnar-Gabor, Dora; Hendrickx, Johann; Tran, Vinh; Hederos, Markus; Dekany, Gyula; Tellier, Charles
Design of an α-l-transfucosidase for the synthesis of fucosylated HMOs Article de journal
Dans: Glycobiology, vol. 26, no. 3, p. 261–269, 2015, ISSN: 14602423.
@article{Saumonneau2015a,
title = {Design of an α-l-transfucosidase for the synthesis of fucosylated HMOs},
author = {Amélie Saumonneau and Elise Champion and Pauline Peltier-Pain and Dora Molnar-Gabor and Johann Hendrickx and Vinh Tran and Markus Hederos and Gyula Dekany and Charles Tellier},
doi = {10.1093/glycob/cwv099},
issn = {14602423},
year = {2015},
date = {2015-01-01},
journal = {Glycobiology},
volume = {26},
number = {3},
pages = {261--269},
abstract = {Human milk oligosaccharides (HMOs) are recognized as benefiting breast-fed infants in multiple ways. As a result, there is growing interest in the synthesis of HMOs mimicking their natural diversity. Most HMOs are fucosylated oligosaccharides. α-l-Fucosidases catalyze the hydrolysis of α-l-fucose from the non-reducing end of a glucan. They fall into the glycoside hydrolase GH29 and GH95 families. The GH29 family fucosidases display a classic retaining mechanism and are good candidates for transfucosidase activity. We recently demonstrated that the α-l-fucosidase from Thermotoga maritima (TmαFuc) from the GH29 family can be evolved into an efficient transfucosidase by directed evolution (Osanjo et al. 2007). In this work, we developed semi-rational approaches to design an α-l-transfucosidase starting with the α-l-fucosidase from commensal bacteria Bifidobacterium longum subsp. infantis (BiAfcB, Blon-2336). Efficient fucosylation was obtained with enzyme mutants (L321P-BiAfcB and F34I/L321P-BiAfcB) enabling in vitro synthesis of lactodifucotetraose, lacto-N-fucopentaose II, lacto-N-fucopentaose III and lacto-N-difucohexaose I. The enzymes also generated more complex HMOs like fucosylated para-lacto-N-neohexaose (F-p-LNnH) and mono- or difucosylated lacto-N-neohexaose (F-LNnH-I, F-LNnH-II and DF-LNnH). It is worth noting that mutation at these two positions did not result in a strong decrease in the overall activity of the enzyme, which makes these variants interesting candidates for large-scale transfucosylation reactions. For the first time, this work provides an efficient enzymatic method to synthesize the majority of fucosylated HMOs.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Craveur, Pierrick; Joseph, Agnel P; Esque, Jeremy; Narwani, Tarun J; Noël, Floriane; Shinada, Nicolas; Goguet, Matthieu; Leonard, Sylvain; Poulain, Pierre; Bertrand, Olivier; Faure, Guilhem; Rebehmed, Joseph; Ghozlane, Amine; Swapna, Lakshmipuram S; Bhaskara, Ramachandra M; Barnoud, Jonathan; Téletchéa, Stéphane; Jallu, Vincent; Cerny, Jiri; Schneider, Bohdan; Etchebest, Catherine; Srinivasan, Narayanaswamy; Gelly, Jean Christophe; de Brevern, Alexandre G
Protein flexibility in the light of structural alphabets Article de journal
Dans: Frontiers in Molecular Biosciences, vol. 2, no. MAY, p. 1–20, 2015, ISSN: 2296889X.
@article{Craveur2015,
title = {Protein flexibility in the light of structural alphabets},
author = {Pierrick Craveur and Agnel P Joseph and Jeremy Esque and Tarun J Narwani and Floriane Noël and Nicolas Shinada and Matthieu Goguet and Sylvain Leonard and Pierre Poulain and Olivier Bertrand and Guilhem Faure and Joseph Rebehmed and Amine Ghozlane and Lakshmipuram S Swapna and Ramachandra M Bhaskara and Jonathan Barnoud and Stéphane Téletchéa and Vincent Jallu and Jiri Cerny and Bohdan Schneider and Catherine Etchebest and Narayanaswamy Srinivasan and Jean Christophe Gelly and Alexandre G de Brevern},
doi = {10.3389/fmolb.2015.00020},
issn = {2296889X},
year = {2015},
date = {2015-01-01},
journal = {Frontiers in Molecular Biosciences},
volume = {2},
number = {MAY},
pages = {1--20},
abstract = {Protein structures are valuable tools to understand protein function. Nonetheless, proteins are often considered as rigid macromolecules while their structures exhibit specific flexibility, which is essential to complete their functions. Analyses of protein structures and dynamics are often performed with a simplified three-state description, i.e., the classical secondary structures. More precise and complete description of protein backbone conformation can be obtained using libraries of small protein fragments that are able to approximate every part of protein structures. These libraries, called structural alphabets (SAs), have been widely used in structure analysis field, from definition of ligand binding sites to superimposition of protein structures. SAs are also well suited to analyze the dynamics of protein structures. Here, we review innovative approaches that investigate protein flexibility based on SAs description. Coupled to various sources of experimental data (e.g., B-factor) and computational methodology (e.g., Molecular Dynamic simulation), SAs turn out to be powerful tools to analyze protein dynamics, e.g., to examine allosteric mechanisms in large set of structures in complexes, to identify order/disorder transition. SAs were also shown to be quite efficient to predict protein flexibility from amino-acid sequence. Finally, in this review, we exemplify the interest of SAs for studying flexibility with different cases of proteins implicated in pathologies and diseases.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Fontaine, Nicolas; Grondin-perez, Brigitte; Cadet, Frédéric; Offmann, Bernard; Fontaine, Nicolas; Grondin-perez, Brigitte; Cadet, Frédéric; Offmann, Bernard; Fontaine, Nicolas; Grondin-perez, Brigitte; Cadet, Frédéric; Offmann, Bernard
Modeling of a Cell-Free Synthetic System for Biohydrogen Production Article de journal
Dans: Journal of Computer Science & Systems Biology, vol. 8, no. 3, 2015, ISSN: 09747230.
@article{Fontaine2015,
title = {Modeling of a Cell-Free Synthetic System for Biohydrogen Production},
author = {Nicolas Fontaine and Brigitte Grondin-perez and Frédéric Cadet and Bernard Offmann and Nicolas Fontaine and Brigitte Grondin-perez and Frédéric Cadet and Bernard Offmann and Nicolas Fontaine and Brigitte Grondin-perez and Frédéric Cadet and Bernard Offmann},
doi = {10.4172/jcsb.1000181},
issn = {09747230},
year = {2015},
date = {2015-01-01},
journal = {Journal of Computer Science & Systems Biology},
volume = {8},
number = {3},
abstract = {Hydrogen is a good candidate for the next generation fuel with a high energy density and an environment friendly behavior in the energy production phase. Micro-organism based biological production of hydrogen currently suffers low hydrogen production yields because the living cells must sustain different cellular activities other than the hydrogen production to survive. To circumvent this, teams have explored the synthetic assembly of enzymes in-vitro in cell-free systems with specific functions. Such a synthetic cell-free system was recently devised by combining 13 different enzymes to synthesize hydrogen from cellulose or cellobiose with better yield than microorganism-based systems. We used methods based on differential equations calculations to investigate how the initial conditions and the kinetic parameters of the enzymes influenced the productivity of a such system and, through simulations, identified those conditions that would optimize hydrogen production starting with cellobiose as substrate. Further, if the kinetic parameters of the component enzymes of such a system are not known, we showed how, using artificial neural network, it is possible to identify alternative models that account for the rate of production of hydrogen. This work demonstrates how modeling can help in designing and characterizing cell-free systems in synthetic biology. A web-based simulator implementing our differential equations based model is provided freely as a service for non- commercial usage at http://www.bo-protscience.fr/h2.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Le-Bail, Patricia; Lorentz, C; Pencreac'h, G; Soultani-Vigneron, S; Pontoire, B; Giraldo, J. López L; Villeneuve, P; Hendrickx, Johann; Tran, Vinh
Trapping by amylose of the aliphatic chain grafted onto chlorogenic acid: Importance of the graft position Article de journal
Dans: Carbohydrate Polymers, vol. 117, p. 910–916, 2015, ISSN: 01448617.
@article{Le-Bail2015,
title = {Trapping by amylose of the aliphatic chain grafted onto chlorogenic acid: Importance of the graft position},
author = {Patricia Le-Bail and C Lorentz and G Pencreac'h and S Soultani-Vigneron and B Pontoire and J.López L Giraldo and P Villeneuve and Johann Hendrickx and Vinh Tran},
url = {http://dx.doi.org/10.1016/j.carbpol.2014.10.029},
doi = {10.1016/j.carbpol.2014.10.029},
issn = {01448617},
year = {2015},
date = {2015-01-01},
journal = {Carbohydrate Polymers},
volume = {117},
pages = {910--916},
publisher = {Elsevier Ltd.},
abstract = {5-Caffeoylquinic acid (chlorogenic acid), is classified in acid-phenols family and as polyphenolic compounds it possesses antioxidant activity. The oxydative modification of chlorogenic acid in foods may lead to alteration of their qualities; to counteract these degradation effects, molecular encapsulation was used to protect chlorogenic acid. Amylose can interact strongly with a number of small molecules, including lipids. In order to enable chlorogenic acid complexation by amylose, a C16 aliphatic chain was previously grafted onto the cycle of quinic acid. This work showed that for the two lipophilic derivatives of chlorogenic acid: hexadecyl chlorogenate obtained by alkylation and 3-O-palmitoyl chlorogenic acid obtained by acylation; only the 3-O-palmitoyl chlorogenic acid complexed amylose. The chlorogenic acid derivatives were studied by X-ray diffraction, differential scanning calorimetry and NMR to elucidate the interaction. By comparing the results with previous work on the complexation of amylose by 4-O-palmitoyl chlorogenic acid, the importance of the aliphatic chain position on the cycle of the quinic acid is clearly highlighted. A study in molecular modeling helped to understand the difference in behavior relative to amylose of these three derivatives of chlorogenic acid.},
keywords = {},
pubstate = {published},
tppubtype = {article}
}
Sanejouand, Yves-Henri
Simplified flexibility analysis of proteins Chapitre d'ouvrage
Dans: Fuxreiter, Monika (Ed.): Computational Approaches to Protein Dynamics: From Quantum to Coarse-Grained Methods, Chapitre 5, p. 153–182, CRC Press, 2015.
@inbook{EQ1:SANEJOUAND:2015,
title = {Simplified flexibility analysis of proteins},
author = {Yves-Henri Sanejouand},
editor = {Monika Fuxreiter},
url = {http://arxiv.org/abs/1312.5639},
year = {2015},
date = {2015-01-01},
booktitle = {Computational Approaches to Protein Dynamics: From Quantum to Coarse-Grained Methods},
pages = {153--182},
publisher = {CRC Press},
chapter = {5},
abstract = {A simple way to get insights about the possible functional motions of a protein is to perform a normal mode analysis (NMA). Indeed, it has been shown that low-frequency modes thus obtained are often closely related to domain motions involved in protein function. Moreover, because protein low-frequency modes are known to be robust, NMA can be performed using coarse-grained models. As a consequence, it can be done for large ensembles of conformations as well as for large systems, like the ribosome, whole virus capsids, etc. Unexpectedly, on the high-frequency side, modes obtained with cutoff-based coarse-grained models also seem able to provide useful insights on protein dynamical properties.},
keywords = {},
pubstate = {published},
tppubtype = {inbook}
}












